決定木の分割の考え方
決定木のデータを特性量によって分割するには、分割後のノードの状態ができるだけうまく分かれていることが必要となる。この「うまく分かれている」状態は、言い換えれば分割後のノード内のデータができるだけ「揃っている」ともいえる。たとえば2クラスの分類をする場合、ノード内に1つのクラスしか含まれていない場合は最も「純度が高い」状態であり、2つのクラスのデータが半分ずつ含まれている場合に最も「純度が低い」状態となる。
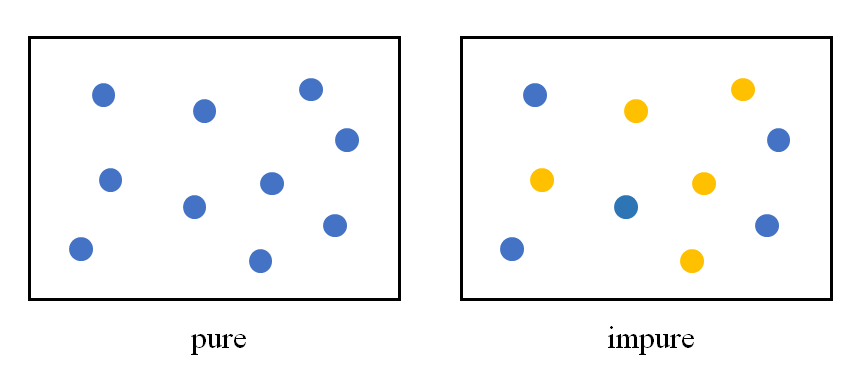
このような状態を定量的に表すのにエントロピーとジニ不純度という2つの考え方があるが、ここではそれらを確認する。
エントロピー(平均情報量)とジニ不純度
定義
クラスc = 1~Cに属するデータがノードtに属しており、各クラスごとのデータ数をnc、データの総数をNとする。この場合、このノードの純度/不純度を表すのに、エントロピー(entropy、平均情報量)とジニ不純度(Gini impurity)という2つの考え方がある。ノードtのエントロピーをIH(t)、ジニ不純度をIG(t)と表すと、それぞれの定義は以下の通り。
(1) 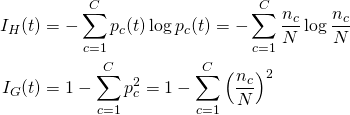
エントロピーの対数の底は何でもいいが、分類するクラス数にすると最も高いエントロピーが1になって都合がよいようだ)。
ジニ不純度については次の表現の方が直感的にわかりやすい。
(2) 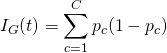
これを展開すると先のIGと同じ形になるが、この形だと関数形が上に凸でpc = 0, 1でIG = 0となることがわかる。
計算例
クラス数Cのデータについて、あるノード内のデータが全て同じクラスの場合、純度が高い/不純度が低い。
(3) 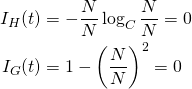
ノード内で全てのクラスのデータが同じ数ずつある場合、純度が低い/不純度が高い。
(4) 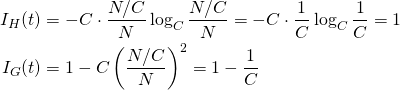
分布
あるノード内にN個の2クラスデータがあり、クラス1のデータ数をnとする。このとき、クラス1のデータの発生率pに対するエントロピー、ジニ不純度の分布は以下のようになる。
(5) 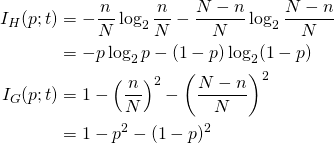
これらをグラフ化したのが以下の図。p = 0.5で双方最大値をとり、エントロピーは1 、ジニ不純度は0.5。グラフの形状を比較するため、ジニ不純度を2倍した線も入れている。
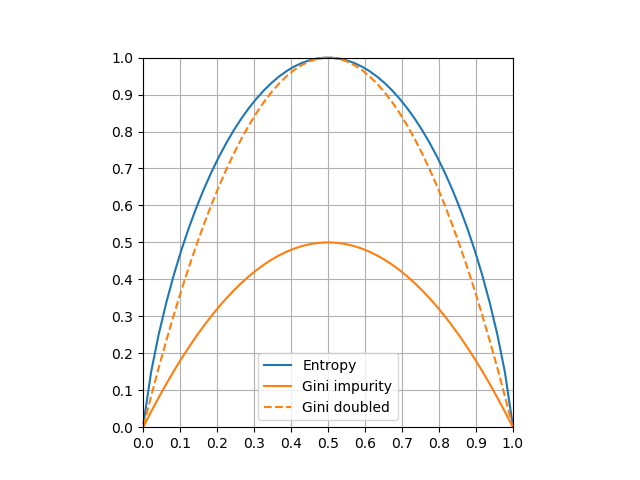
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 |
import numpy as np import matplotlib.pyplot as plt eps = 1e-3 p = np.linspace(eps, 1 - eps) Ih = -p * np.log2(p) - (1 - p) * np.log2(1 - p) Ig = 1 - p**2 - (1 - p)**2 fig, ax = plt.subplots() ax.plot(p, Ih, c='tab:blue', label="Entropy", clip_on=False) ax.plot(p, Ig, c='tab:orange', label="Gini impurity") ax.plot(p, Ig*2, c='tab:orange', ls='dashed', label="Gini doubled" , clip_on=False) ax.set_xlim(0, 1) ax.set_ylim(0, 1) ax.set_xticks(np.linspace(0, 1, 11)) ax.set_yticks(np.linspace(0, 1, 11)) ax.grid(True) ax.set_aspect('equal') ax.legend() plt.show() |
ノード分割の考え方~利得
親ノードを子ノードに分割するのに最も妥当な分割とするには、分割後の子ノードの純度ができるだけ高くなるような特徴量を探すことになる。ある特徴量について、親ノードから子ノードに分割したときにどれだけ純度が高くなったかを比較する量として、利得(情報利得、gain)が用いられる。
親ノードtPにc = 1~Cのクラスのデータがそれぞれncずつあるとする。このときの親ノードのエントロピーIH(tP)、ジニ不純度IG(tP)は式(1)で計算される。
まずエントロピーについて考える。ある特徴量fを定めると左右のノードのデータ分布が決まり、その時の左ノードtL、右ノードtLのエントロピーが以下のように計算される。
(6) 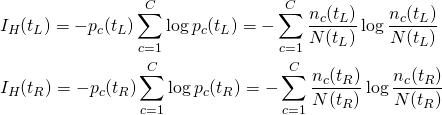
このとき、それぞれのノードのエントロピーにノードの重みwL, wRを掛け、これを親ノードのエントロピーから引いた量を利得(gain、情報利得)という。重みを各ノードのデータ数の比率とすると、利得は以下のように計算される。
(7) 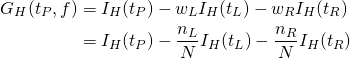
ジニ不純度についても同様の計算ができる。
(8) 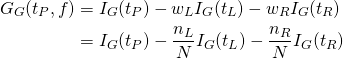
利得は、ある特徴量の値によって分割した後の状態が、分割前の状態に対してどれだけ純度が高くなったかを表す。
決定木のノードを分割するにあたっては、子ノードの純度ができるだけ高くなるように(エントロピー/ジニ不純度が小さくなるように)fを選ぶことになる。
簡単な例
特徴量が1つでデータ数が少ないケースで、利得の計算を確認してみる。
000111と並んでいる場合
クラス0、1がこのように並んでいるとき、左から境界を動かしていったときの左右のノードの不純度、利得について計算した結果は以下の通り。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 |
IHP=1.0, IGP=0.5 [0][0 0 1 1 1]: IHL=-0.000, IHR= 0.971, G_H= 0.191 IGL= 0.000, IGR= 0.480, G_G= 0.100 [0 0][0 1 1 1]: IHL=-0.000, IHR= 0.811, G_H= 0.459 IGL= 0.000, IGR= 0.375, G_G= 0.250 [0 0 0][1 1 1]: IHL=-0.000, IHR= 0.000, G_H= 1.000 IGL= 0.000, IGR= 0.000, G_G= 0.500 [0 0 0 1][1 1]: IHL= 0.811, IHR= 0.000, G_H= 0.459 IGL= 0.375, IGR= 0.000, G_G= 0.250 [0 0 0 1 1][1]: IHL= 0.971, IHR= 0.000, G_H= 0.191 IGL= 0.480, IGR= 0.000, G_G= 0.100 |
この場合、当然のことながら真ん中でノードを分割することで2つのクラスがきれいに分かれ、利得もこれを表している。
00100111と並んでいる場合
今度は一部に他のクラスが紛れ込んでいる場合。左のクラス0の集団に1つだけクラス1のデータが含まれているときの挙動を確認する。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 |
IHP=1.0, IGP=0.5 [0][0 1 0 0 1 1 1]: IHL=-0.000, IHR= 0.985, G_H= 0.138 IGL= 0.000, IGR= 0.490, G_G= 0.071 [0 0][1 0 0 1 1 1]: IHL=-0.000, IHR= 0.918, G_H= 0.311 IGL= 0.000, IGR= 0.444, G_G= 0.167 [0 0 1][0 0 1 1 1]: IHL= 0.918, IHR= 0.971, G_H= 0.049 IGL= 0.444, IGR= 0.480, G_G= 0.033 [0 0 1 0][0 1 1 1]: IHL= 0.811, IHR= 0.811, G_H= 0.189 IGL= 0.375, IGR= 0.375, G_G= 0.125 [0 0 1 0 0][1 1 1]: IHL= 0.722, IHR= 0.000, G_H= 0.549 IGL= 0.320, IGR= 0.000, G_G= 0.300 [0 0 1 0 0 1][1 1]: IHL= 0.918, IHR= 0.000, G_H= 0.311 IGL= 0.444, IGR= 0.000, G_G= 0.167 [0 0 1 0 0 1 1][1]: IHL= 0.985, IHR= 0.000, G_H= 0.138 IGL= 0.490, IGR= 0.000, G_G= 0.071 |
左から1つ目2つ目と境界を動かしていくと少しずつ利得が上昇するが、左側のノードにクラス1のデータが入ってきたところでその不純度が跳ね上がり、利得が下がる(8~10行目)。その後再び利得は上昇し、右側のデータがクラス1のみ3つとなった時に利得が最大となっている。このとき左側のノードにクラス1のデータが1つ含まれているが、他の4つのデータがクラスゼロと多いため、不純度は比較的低い。
利得が最大となる時でも、完全にクラスが分かれた時に比べて半分近くの利得だが、これはデータ数の多い左側に異なるクラスのデータが含まれているからと考えられる。
ここまでの計算に使ったコードは以下の通り。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 |
import numpy as np def impurity(node): N = node.size n0 = node[node==0].size n1 = node[node==1].size p0, p1 = n0 / N, n1 / N entropy = - (p0 * np.log2(p0) if p0 > 0 else 0) \ - (p1 * np.log2(p1) if p1 > 0 else 0) gini = 1 - p0*p0 - p1*p1 return entropy, gini #node = np.array([0, 0, 0, 1, 1, 1]) node = np.array([0, 0, 1, 0, 0, 1, 1, 1]) IHP, IGP = impurity(node) print("IHP={}, IGP={}".format(IHP, IGP)) for pos in range(1, node.size): node_left = node[0:pos] node_right = node[pos:node.size] node_text = "{}{}".format(node_left, node_right) IHL, IGL = impurity(node_left) IHR, IGR = impurity(node_right) wL = node_left.size / node.size wR = node_right.size / node.size GH = IHP - wL*IHL - wR*IHR GG = IGP - wL*IGL - wR*IGR print("{}:".format(node_text)) print(" IHL={:6.3f}, IHR={:6.3f}, G_H={:6.3f}".format(IHL, IHR, GH)) print(" IGL={:6.3f}, IGR={:6.3f}, G_G={:6.3f}".format(IGL, IGR, GG)) |