概要
主成分分析(PCA)において、次元削減により主成分の一部だけを残し、それを逆変換することを考える。
結論から言うと、以下の2つの操作は同じ結果をもたらす。
- 全ての主成分を用いて変換し、削減する主成分に対応する元の特徴量を0とし、逆変換する
- 削減する主成分より低次の主成分のみで変換し、それを逆変換する
簡単な例
全主成分を使った手順
概要
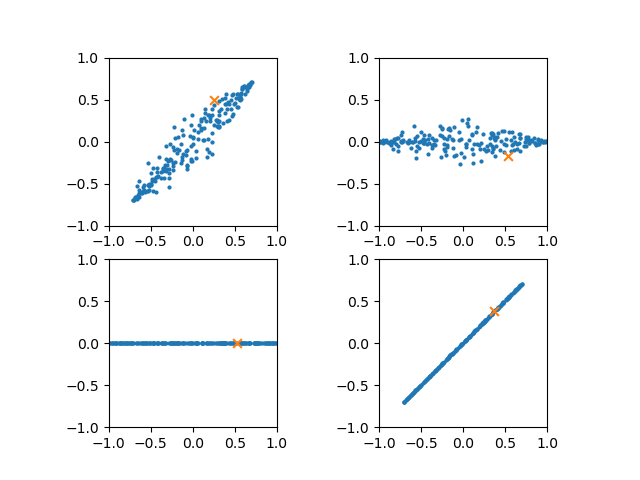
最初の例は、2次元のデータについて以下のような操作を行っている。
- 2つの主成分まで使ってデータを変換
- 第2主成分に対応する変換後のデータを0にする
- そのデータを逆変換する
元データの作成
まず元データを作成し、左上に散布図を描画。
元データは水平線にcosine状に正規分布するノイズを乗せ、それを45度回転させている。
また、適当な位置に特定の点を1つ定義している。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 |
import numpy as np import matplotlib.pyplot as plt from sklearn.decomposition import PCA np.random.seed(0) n_data = 200 cs = 1 / np.sqrt(2) x = np.linspace(-1, 1, n_data) y = np.random.randn(n_data) * np.cos(x * np.pi / 2) / 6 x, y = cs * x - cs * y, cs * x + cs * y xp, yp = 0.25, 0.5 X = np.array([x, y]).reshape(-1, 2) Xp = np.array([xp, yp]).reshape(-1, 2) fig1, axes1 = plt.subplots(2, 2) axes1[0, 0].scatter(X[:, 0], X[:, 1], marker='o', s=4) axes1[0, 0].scatter(Xp[0, 0], Xp[0, 1], marker='x', s=40) print("Xp original:{}".format(Xp)) # Xp original:[[0.25 0.5 ]] ..... for axis in axes1.ravel(): axis.set_aspect('equal') axis.set_xlim(-1, 1) axis.set_ylim(-1, 1) plt.show() |
フィッティングと元データの変換
次に元データをPCAによって変換し、変換後の散布図を右上に描画。
斜めだった分布が、第1主成分がx軸と重るように変換されて水平になる。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 |
..... pca = PCA().fit(X) X_trans = pca.transform(X) Xp_trans = pca.transform(Xp) axes1[0, 1].scatter(X_trans[:, 0], X_trans[:, 1], marker='o', s=4) axes1[0, 1].scatter(Xp_trans[0, 0], Xp_trans[0, 1], marker='x', s=40) print("Xp transfomed:{}".format(Xp_trans)) # Xp transfomed:[[ 0.53055026 -0.17107022]] ..... |
第2主成分のデータの削除
変換後のデータにおいて、第2主成分に関する値を0とし、左下に散布図を描画。
第2成分に相当する垂直成分が0になる。
|
1 2 3 4 5 6 7 8 |
..... X_trans[:, 1] = 0 Xp_trans[0, 1] = 0 axes1[1, 0].scatter(X_trans[:, 0], X_trans[:, 1], marker='o', s=4) axes1[1, 0].scatter(Xp_trans[0, 0], Xp_trans[0, 1], marker='x', s=40) ..... |
逆変換
第2主成分を0としたデータを逆変換して描画。
第1主成分に直角な第2主成分が0となり、全点が一直線上に並ぶ。
|
1 2 3 4 5 6 7 8 9 |
X_inv = pca.inverse_transform(X_trans) Xp_inv = pca.inverse_transform(Xp_trans) axes1[1, 1].scatter(X_inv[:, 0], X_inv[:, 1], marker='o', s=4) axes1[1, 1].scatter(Xp_inv[0, 0], Xp_inv[0, 1], marker='x', s=40) print("Xp inversed:{}".format(Xp_inv)) # Xp inversed:[[0.37112019 0.37919056]] ..... |
最初から主成分を限定する手順
概要
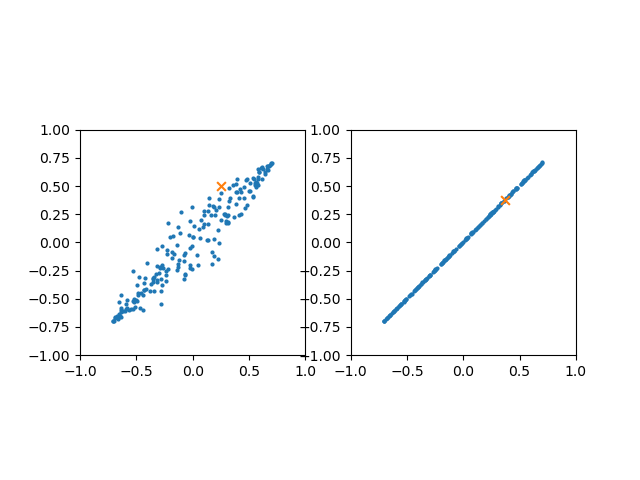
次の例は元の特徴量を操作せず、以下のような手順に寄っている。
- PCAのモデル生成時に、
n_component=1とする - そのPCAモデルで元データを変換する
- 変換したデータを逆変換する
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 |
fig2, axes2 = plt.subplots(1, 2) axes2[0].scatter(X[:, 0], X[:, 1], marker='o', s=4) axes2[0].scatter(Xp[0, 0], Xp[0, 1], marker='x', s=40) print("Xp original:{}".format(Xp)) # Xp original:[[0.25 0.5 ]] pca = PCA(n_components=1).fit(X) X_trans = pca.transform(X) Xp_trans = pca.transform(Xp) print("Xp transfomed:{}".format(Xp_trans)) # Xp transfomed:[[0.53055026]] X_inv = pca.inverse_transform(X_trans) Xp_inv = pca.inverse_transform(Xp_trans) axes2[1].scatter(X_inv[:, 0], X_inv[:, 1], marker='o', s=4) axes2[1].scatter(Xp_inv[0, 0], Xp_inv[0, 1], marker='x', s=40) print("Xp inversed:{}".format(Xp_inv)) # Xp inversed:[[0.37112019 0.37919056]] for axis in axes2.ravel(): axis.set_aspect('equal') axis.set_xlim(-1, 1) axis.set_ylim(-1, 1) plt.show() |
まとめ
途中で表示させているXpのデータが、2つの手順で全く同じであることがわかる。
最初から主成分を限定することで、元の特徴量を意識せずに次元削減ができる。