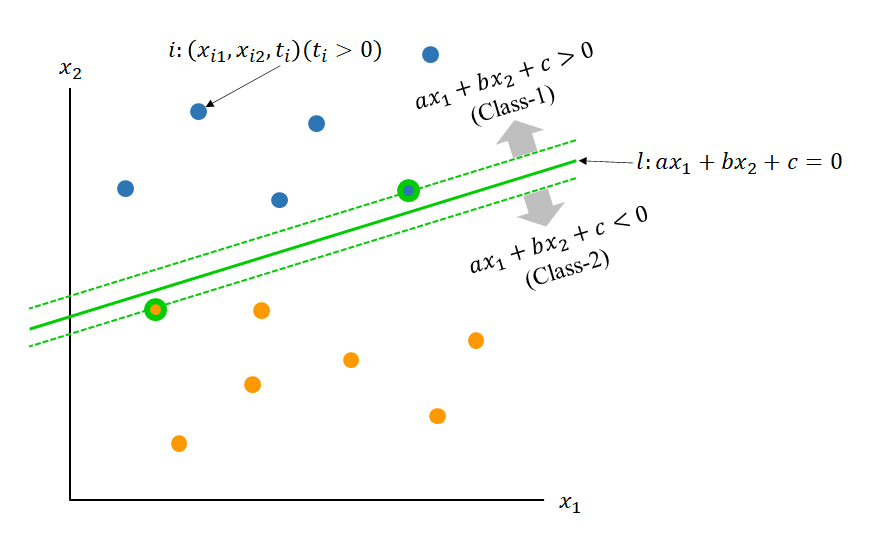
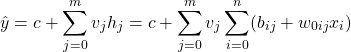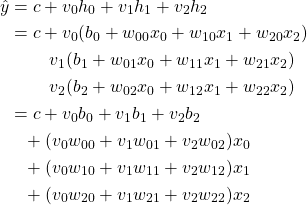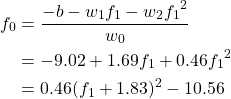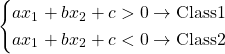概要
書籍”Pythonではじめる機械学習”の2.3.7 カーネル法を用いた”サポートベクタマシン”の写経
線形特徴量の非線形化
線形モデルでは分離不可能なデータ
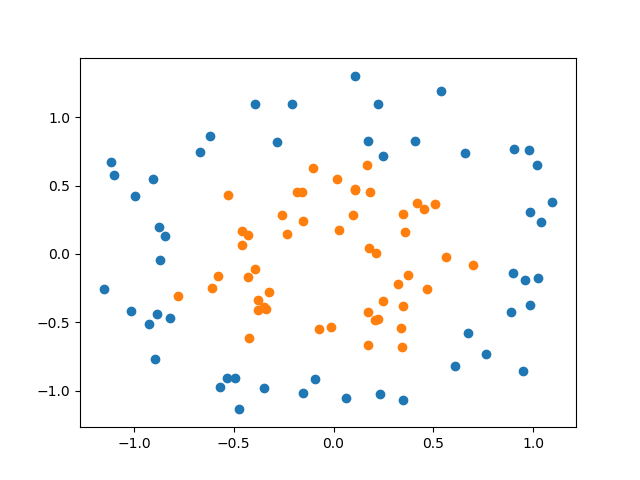
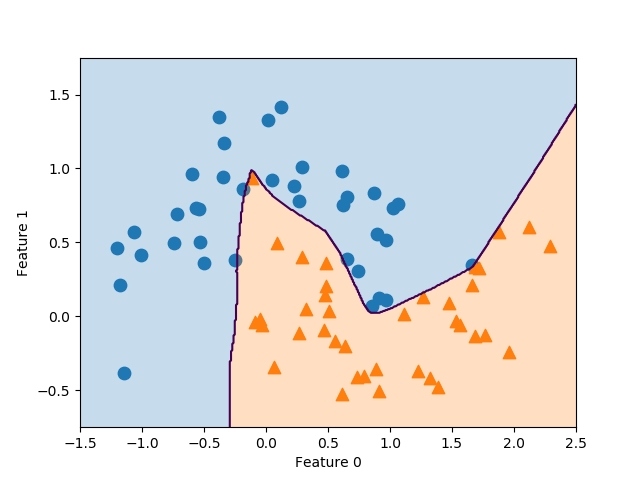
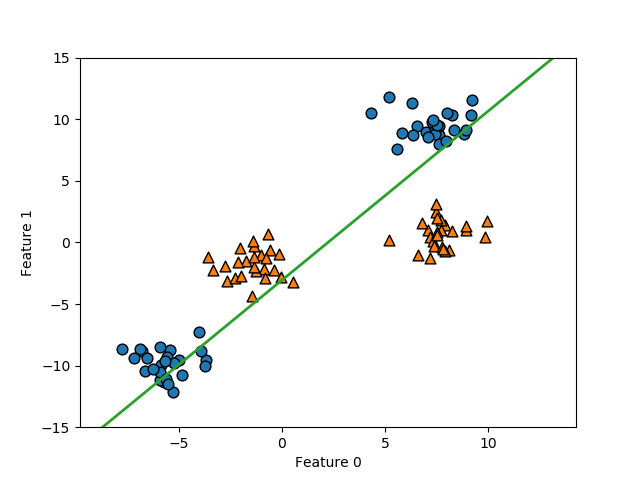
以下は、scikit-learnのmake_blobs()により生成した2特徴量、2クラス分類のデータに線形サポートベクターマシンを適用した例。このとき、決定境界は以下のように得られる。
(1) 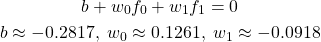
決定境界より上側では多項式の値は負となり、下側では正となるが、この境界は明らかに2つのクラスを分割していない。このように単純な例でも、線形モデルでは的確なクラス分類はできない。
以下のコードでは、原典と以下が異なっている。
- 収束しないという警告を受けて、
LinearSVCのmax_iterをデフォルトの1000より大きな値としている
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 |
import matplotlib.pyplot as plt from sklearn.datasets import make_blobs from sklearn.svm import LinearSVC X, y = make_blobs(centers=4, random_state=8) y = y % 2 linsvm = LinearSVC(max_iter=5500).fit(X, y) y_min, y_max = -15, 15 b = linsvm.intercept_ w0 = linsvm.coef_[0][0] w1 = linsvm.coef_[0][1] x_lower = -(b + w1 * y_min) / w0 x_upper = -(b + w1 * y_max) / w0 fig, ax = plt.subplots() X0 = X[y==0] X1 = X[y==1] ax.scatter(X0[:, 0], X0[:, 1], marker='o', s=60, ec='k') ax.scatter(X1[:, 0], X1[:, 1], marker='^', s=60, ec='k') ax.plot([x_lower, x_upper], [y_min, y_max], linewidth=2, c='tab:green') ax.set_ylim(y_min, y_max) ax.set_xlabel("Feature-0") ax.set_ylabel("Feature-1") plt.show() |
非線形特徴量の追加
ここで、特徴量1の2乗を新たな特徴量として加える。この場合、3つの特徴量に対して3次元空間内に各点が位置し、それぞれがクラス0/1に属している。新たな特徴量の追加によって、その軸の方向に各点が立ち上がり、真ん中の三角形の点群と両側の丸印の点群が平面でうまく分割できそうである。
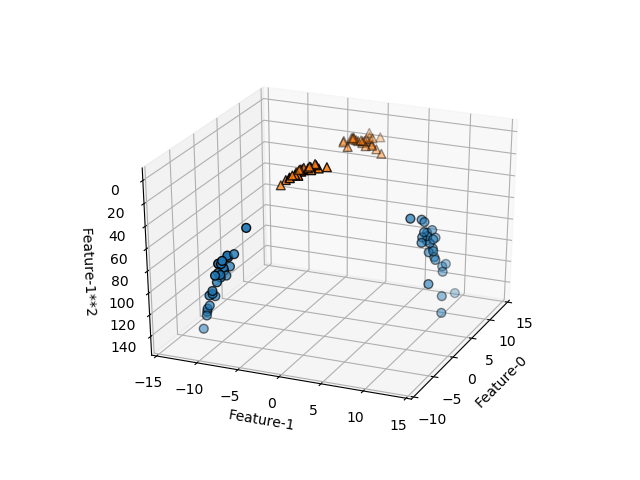
このデータセットに対して、線形SVMを適用し、決定境界を描いたのが以下の画像。特徴量が2つの場合の決定境界は直線だったが、特徴が3つになると決定境界は平面となる。予想通り、単純な平面で2つのクラスが分けられている。この決定境界の式は以下のようになる。
(2) 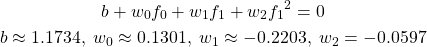
この平面に対して上側(f12が小さい側)では多項式の値は正となり、その反対側では負となる。
以下のコードは、原典と以下が異なっている。
- 収束しないという警告を受けて、
LinearSVCのmax_iterをデフォルトの1000より大きな値としている
Axes3Dの生成の仕方を最新のバージョンに合ったものとしている
- 原典では
Figureオブジェクトを生成し、それをビューに関する引数とともにAxes3Dコンストラクターに渡している
- 本コードでは、
subplotsの引数でprojectionを指定してFigureとAxes3Dを同時に生成し、veiw_init()でビューに関する引数を指定
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 |
import numpy as np import matplotlib.pyplot as plt from sklearn.datasets import make_blobs from sklearn.svm import LinearSVC from mpl_toolkits.mplot3d import Axes3D X, y = make_blobs(centers=4, random_state=8) y = y % 2 X_new = np.hstack((X, X[:, 1].reshape(-1, 1)**2)) X0, X1 = X_new[y==0], X_new[y==1] linsvc = LinearSVC(max_iter=3700).fit(X_new, y) intercept = linsvc.intercept_ coef = linsvc.coef_.ravel() fig, ax = plt.subplots(subplot_kw=dict(projection='3d')) ax.view_init(elev=-152, azim=-23) u = np.linspace(X_new[:, 0].min() - 2, X_new[:, 1].max() + 2) v = np.linspace(X_new[:, 0].min() - 2, X_new[:, 1].max() + 2) u, v = np.meshgrid(u, v) w = -(coef[0] * u + coef[1] * v + intercept) / coef[2] ax.scatter(X0[:, 0], X0[:, 1], X0[:, 2], marker='o', s=40, ec='k') ax.scatter(X1[:, 0], X1[:, 1], X1[:, 2], marker='^', s=40, ec='k') ax.plot_wireframe(u, v, w, rstride=8, cstride=8, color='tab:green', alpha=0.5) ax.set_xlabel("Feature-0") ax.set_ylabel("Feature-1") ax.set_zlabel("Feature-1**2") plt.show() |
元の特徴量に対する決定境界
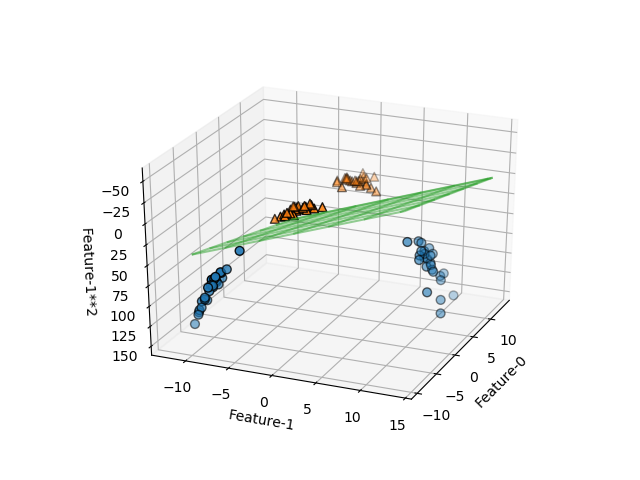
上の例では特徴量は3つだが、最後の特徴量は2つ目の特徴量f1から計算される量であり、実質は2つの特徴量が決まれば決定境界が決まる。3次元空間内の平面の決定境界を以下のように書きなおすと、このことが確認できる。
(3) 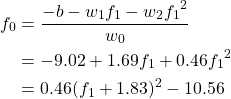
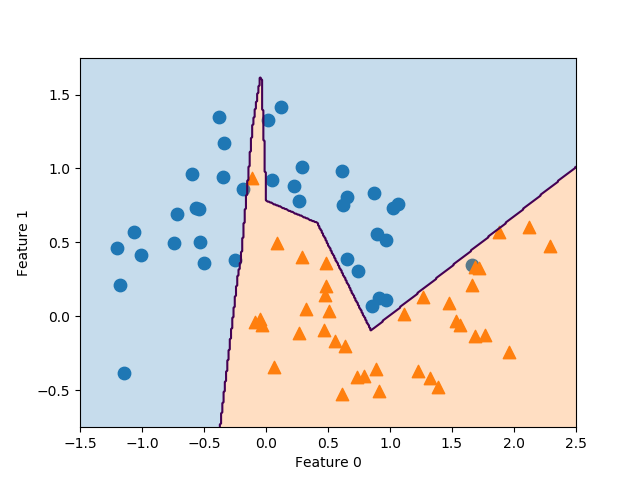
これを2つの特徴量に対する決定境界として描画したのが以下の図で、境界が2次関数となっているのが確認できる。
SVMそのものは線形の決定境界しか得られないが、非線形化した特徴量を追加することによって、より複雑な決定境界とすることができる。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 |
import numpy as np import matplotlib.pyplot as plt from sklearn.datasets import make_blobs from sklearn.svm import LinearSVC from mpl_toolkits.mplot3d import Axes3D X, y = make_blobs(centers=4, random_state=8) y = y % 2 X_new = np.hstack((X, X[:, 1].reshape(-1, 1)**2)) linsvc = LinearSVC(max_iter=3700).fit(X_new, y) intercept = linsvc.intercept_ x_min, x_max = -12, 12 y_min, y_max = -15, 15 u = np.linspace(x_min, x_max, 500) v = np.linspace(y_min, y_max, 500) u, v = np.meshgrid(u, v) w = v**2 decision = linsvc.predict(np.c_[u.ravel(), v.ravel(), w.ravel()]) fig, ax = plt.subplots() ax.scatter(X_new[y==0, 0], X_new[y==0, 1], marker='o', s=60, ec='k') ax.scatter(X_new[y==1, 0], X_new[y==1, 1], marker='^', s=60, ec='k') ax.contourf(u, v, decision.reshape(u.shape), levels=1, colors=['tab:blue', 'tab:orange'], alpha=0.4) ax.contour(u, v, decision.reshape(u.shape), levels=1, colors='k') ax.set_xlim(x_min, x_max) ax.set_ylim(y_min, y_max) ax.set_xlabel("Feature-0") ax.set_ylabel("Feature-1") plt.show() |
カーネルトリック
概要
上記の例では特徴量の1つを2次として新たな特徴量とした。特徴量の非線形化としては、このように特徴量の累乗とするほか、異なる特徴量同士の積を交互作用として導入することが考えられる。ただし、特徴量の数が多くなった時に、それらの全ての組み合わせに対する積を考えると、計算量が膨れ上がる。カーネルトリック(kernel trick)とは、拡張された特徴量空間でのデータ間の距離を、実際の拡張計算をせずに行う方法らしい。
受け売りをそのまま書いておくと、SVMで広く用いられているカーネルトリックのマッピング方法は以下の2つとのこと。
- 多項式カーネル(polynomial kernel):もとの特徴量の特定の次数までの全ての多項式を計算
- 放射既定関数(radial basis function: RBF)カーネルとも呼ばれるガウシアンカーネル:直感的には全次数の全ての多項式を考えるが、次数が高くなるにつれて特徴量の重要性を小さくする
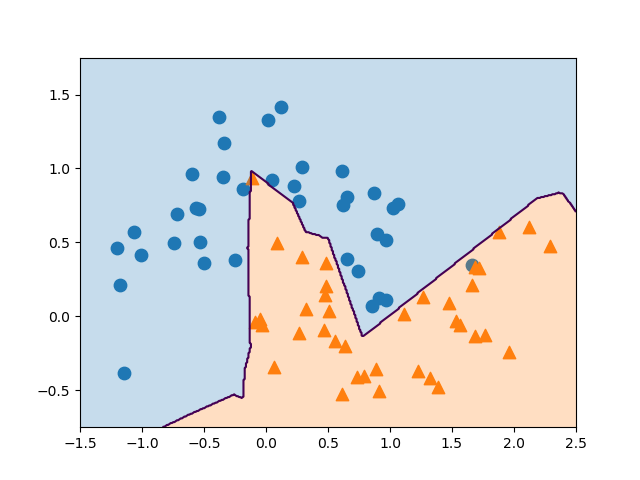
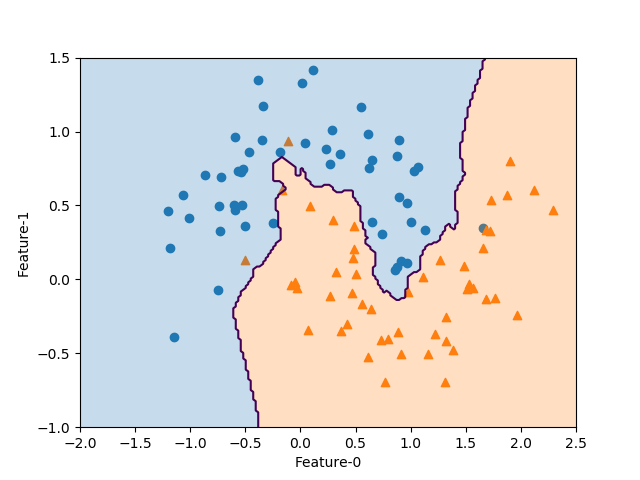
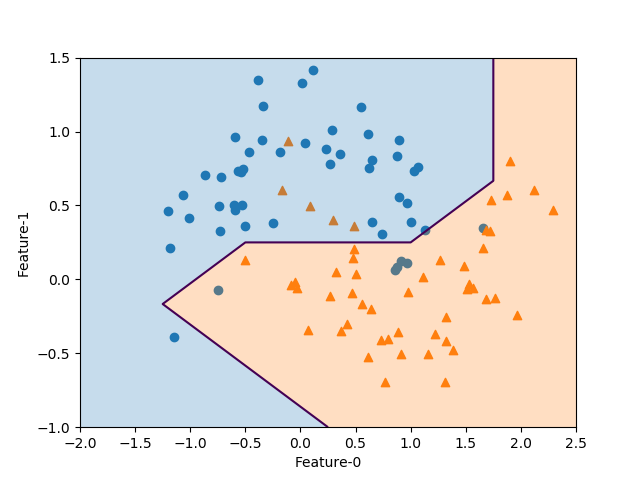
以下はforgeデータセットに対して、カーネルトリックを用いたSVCを適用した例。直線はLinearSVCによる決定境界で、曲線はガウシアンカーネル(RBF)によるSVCの決定境界で、カーネル関数は以下のような形。
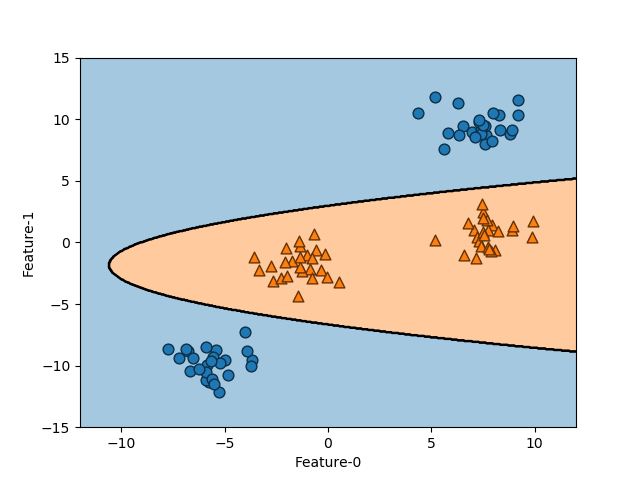
(4) 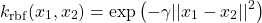
scikit-learnのSVCの引数で、kernel='rbf'、C=10、gamma=0.1と指定している。
線形モデルの決定境界が直線なのに対して、カーネルトリックによる決定境界は、非線形化した特徴量を導入していることから曲線となっている。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 |
import numpy as np import matplotlib.pyplot as plt from sklearn.svm import SVC, LinearSVC X = np.array( \ [[ 9.96346605, 4.59676542], [11.0329545, -0.16816717], [11.54155807, 5.21116083], ..... [ 9.50169345, 1.93824624], [ 9.15072323, 5.49832246], [11.563957, 1.3389402 ]] ) y = np.array( \ [1, 0, 1, 0, 0, 1, 1, 0, 1, 1, 1, 1, 0, 0, 1, 1, 1, 0, 0, 1, 0, 0, 0, 0, 1, 0] ) f0min, f0max = 7.5, 12.5 f1min, f1max = -1, 6 linsvc = LinearSVC(max_iter=4000).fit(X, y) intercept = linsvc.intercept_ coef = linsvc.coef_.ravel() svc = SVC(kernel='rbf', C=10, gamma=0.1).fit(X, y) u = np.linspace(f0min, f0max, 400) v = np.linspace(f1min, f1max, 400) u, v = np.meshgrid(u, v) pred = svc.predict(np.c_[u.ravel(), v.ravel()]).reshape(u.shape) fig, ax = plt.subplots() ax.scatter(X[y==0][:, 0], X[y==0][:, 1], marker='o', s=60, fc='tab:blue', ec='k') ax.scatter(X[y==1][:, 0], X[y==1][:, 1], marker='^', s=60, fc='tab:orange', ec='k') sv_class = y[svc.support_] ax.scatter(svc.support_vectors_[sv_class==0][:, 0], svc.support_vectors_[sv_class==0][:, 1], marker='o', s=150, fc='tab:blue', ec='blue', linewidth=3) ax.scatter(svc.support_vectors_[sv_class==1][:, 0], svc.support_vectors_[sv_class==1][:, 1], marker='^', s=150, fc='tab:orange', ec='red', linewidth=3) f1 = lambda f0: -(intercept + coef[0]*f0) / coef[1] ax.plot([f0min, f0max], [f1(f0min), f1(f0max)]) ax.contour(u, v, pred, levels=[0.5]) ax.set_xlim(f0min, f0max) ax.set_ylim(f1min, f1max) ax.tick_params(bottom=False, left=False, labelbottom=False, labelleft=False) ax.set_xlabel("Feature-0") ax.set_ylabel("Feature-1") plt.show() |
scikit-learnのSVCクラスには、サポートベクターに関する以下のパラメーターがある。
- support_:データセットにおけるサポートベクターのインデックス(1次元配列)
- support_vector_:サポートベクターの配列(2次元配列)
38~44行目で、これらのパラメーターを使ってサポートベクターを強調表示している。
パラメータ調整
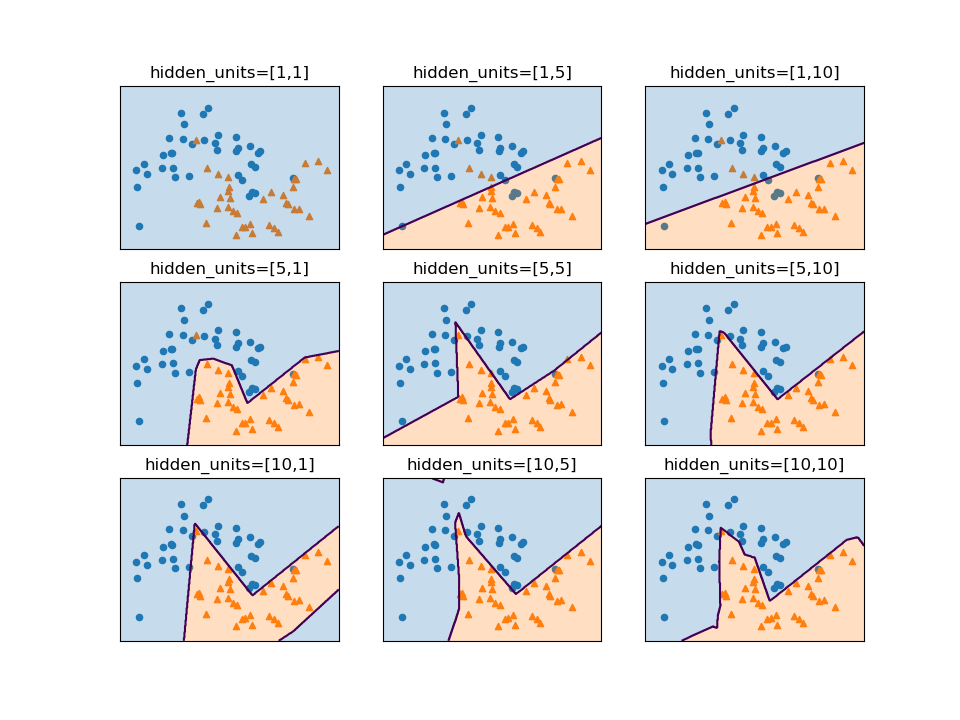
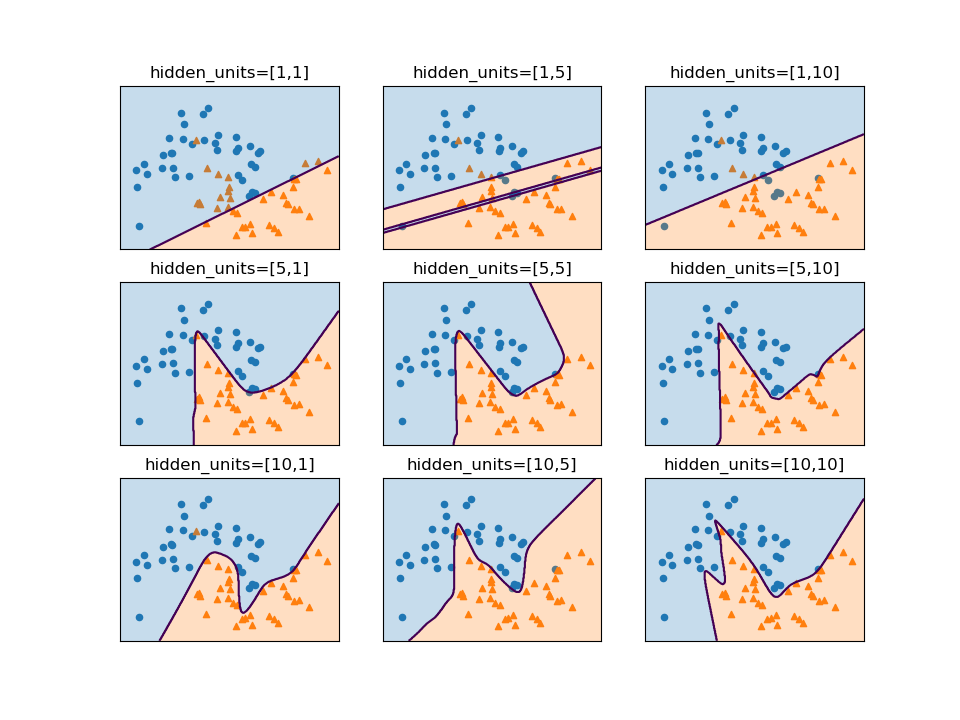
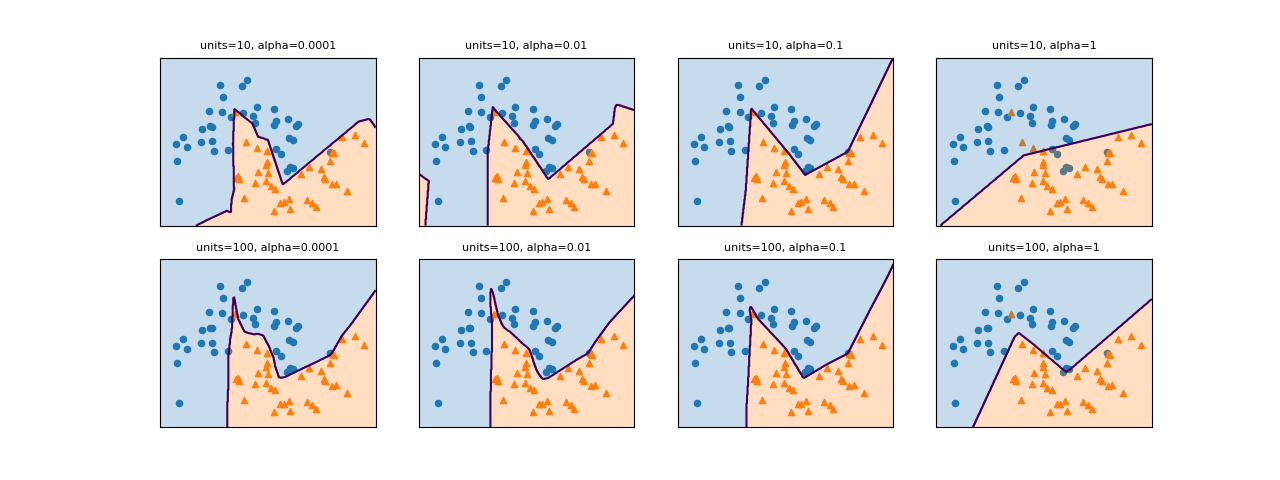
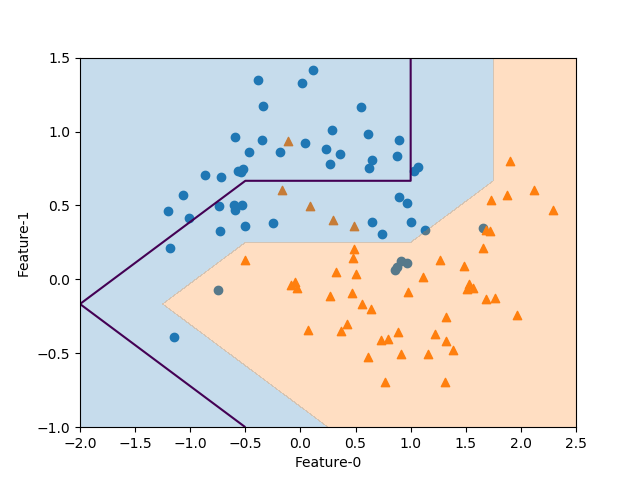
SVCモデルでパラメーターCとgammaの値を変化させたときの決定境界は以下の通り。
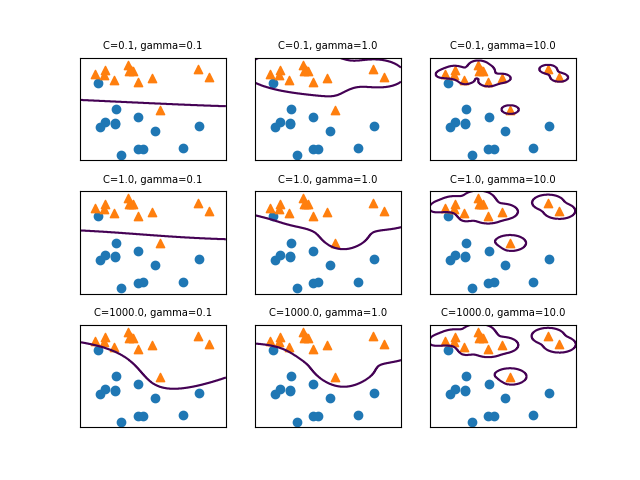
gammaはガウシアンカーネルの直径(σ2に相当)の逆数で、この値が小さいと直径が大きくなり、より多くの点を近いと判断するようになる。左の方はgammaが小さく広域のデータをまとめようとするため、決定境界は大まかとなり、右の方はgammaが大きく近いもの同士をまとめようとする傾向となる。
Cは正則化の強さの逆数で、上の方ほどCの値が小さく正則化が強く効くため、決定境界はよりまっすぐとなり、下の方ほど正則化が弱く個々のデータの影響を受ける。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 |
import numpy as np import matplotlib.pyplot as plt from sklearn.svm import SVC from itertools import product X = np.array( \ [[ 9.96346605, 4.59676542], [11.0329545, -0.16816717], [11.54155807, 5.21116083], ..... [ 9.50169345, 1.93824624], [ 9.15072323, 5.49832246], [11.563957, 1.3389402 ]] ) y = np.array( \ [1, 0, 1, 0, 0, 1, 1, 0, 1, 1, 1, 1, 0, 0, 1, 1, 1, 0, 0, 1, 0, 0, 0, 0, 1, 0] ) f0min, f0max = 7.5, 12.5 f1min, f1max = -1, 6 u = np.linspace(f0min, f0max, 400) v = np.linspace(f1min, f1max, 400) u, v = np.meshgrid(u, v) C_list = [0.1, 1, 1000] gamma_list = [0.1, 1, 10] params = product(C_list, gamma_list) plt.rcParams['font.size'] = 6 fig, axs = plt.subplots(3, 3, figsize=(6.4, 4.8)) axs_1d = axs.ravel() fig.subplots_adjust(hspace=0.3) for ax, param in zip(axs_1d, params): svc = SVC(kernel='rbf', C=param[0], gamma=param[1]).fit(X, y) pred = svc.predict( np.hstack([u.ravel().reshape(-1, 1), v.ravel().reshape(-1, 1)]) ).reshape(u.shape) ax.scatter(X[y==0][:, 0], X[y==0][:, 1], marker='o') ax.scatter(X[y==1][:, 0], X[y==1][:, 1], marker='^') ax.contour(u, v, pred, levels=[0.5]) ax.tick_params(left=False, bottom=False, labelleft=False, labelbottom=False) ax.set_title("C={:.1f}, gamma={:.1f}".format(param[0], param[1])) plt.show() |
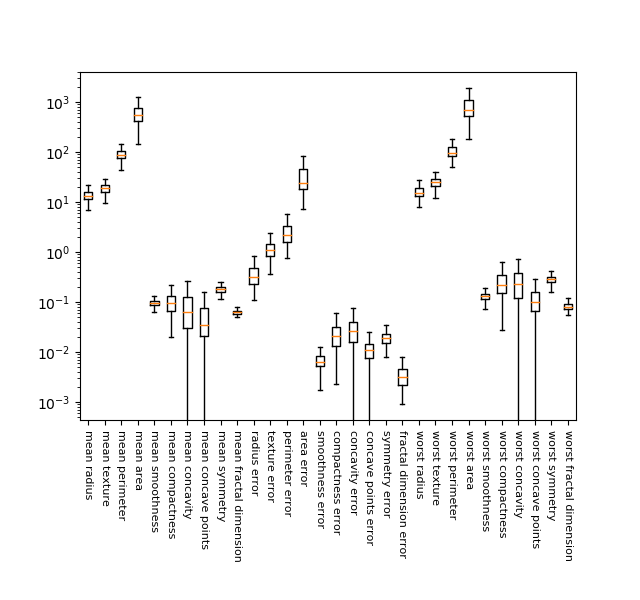
Breast cancerデータへの適用例
Breast cancerデータへの適用例で、特徴量データの大きさやレンジが大きくばらついていること、特徴量データをそのまま使った場合に過学習となること、特徴量データに前処理を施して正規化(normalize)した場合に精度が向上することを示している。
SVMの特徴
SVMの特徴量を受け売りのまままとめておく。
- データにわずかな特徴量しかない場合も複雑な決定境界を生成可能(低次元でも高次元でもうまく機能)
- サンプルの個数が大きくなるとうまく機能しない(10万サンプルくらいになると、実行時間やメモリ使用量の面で難しくなる
- 注意深いデータの前処理とパラメーター調整が必要
- 検証が難しい(予測に対する理由を理解することが難しい)
- RBFの場合、Cやgammaを大きくするとより複雑なモデルになる(2つのパラメーターは強く相関するため、同時に調整する必要がある)
今後の課題~覚え書き
カーネル関数
(5) 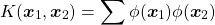
多項式カーネル
(6) 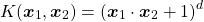
ガウシアンカーネル
(7) 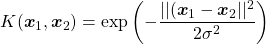
![]()