概要
diabetesデータは、年齢や性別など10個の特徴量と、それらの測定1年後の糖尿病の進行度に関する数値を、442人について集めたデータ。出典は”From Bradley Efron, Trevor Hastie, Iain Johnstone and Robert Tibshirani (2004) “Least Angle Regression,” Annals of Statistics (with discussion), 407-499″。
ここではPythonのscikit-learnにあるdiabetesデータの使い方をまとめる。
データの取得とデータ構造
Pythonで扱う場合、scikit-learn.datasetsモジュールにあるload_diabetes()でデータを取得できる。データはBunchクラスのオブジェクト
|
1 2 3 4 5 6 |
from sklearn.datasets import load_diabetes ds = load_diabetes() for key, value in zip(ds.keys(), ds.values()): print("{}:\n{}\n".format(key, value)) |
データの構造は辞書型で、442人の糖尿病に関する10個の特徴量をレコードとした配列、442人の測定1年後の糖尿病の進行度を示す数値データの配列など。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 |
data: [[ 0.03807591 0.05068012 0.06169621 ... -0.00259226 0.01990842 -0.01764613] [-0.00188202 -0.04464164 -0.05147406 ... -0.03949338 -0.06832974 -0.09220405] [ 0.08529891 0.05068012 0.04445121 ... -0.00259226 0.00286377 -0.02593034] ... [ 0.04170844 0.05068012 -0.01590626 ... -0.01107952 -0.04687948 0.01549073] [-0.04547248 -0.04464164 0.03906215 ... 0.02655962 0.04452837 -0.02593034] [-0.04547248 -0.04464164 -0.0730303 ... -0.03949338 -0.00421986 0.00306441]] target: [151. 75. 141. 206. 135. 97. 138. 63. 110. 310. 101. 69. 179. 185. 118. 171. 166. 144. 97. 168. 68. 49. 68. 245. 184. 202. 137. 85. 131. 283. 129. 59. 341. 87. 65. 102. 265. 276. 252. 90. 100. 55. 61. 92. 259. 53. 190. 142. 75. 142. 155. 225. 59. 104. 182. 128. 52. 37. 170. 170. 61. 144. 52. 128. 71. 163. 150. 97. 160. 178. 48. 270. 202. 111. 85. 42. 170. 200. 252. 113. 143. 51. 52. 210. 65. 141. 55. 134. 42. 111. 98. 164. 48. 96. 90. 162. 150. 279. 92. 83. 128. 102. 302. 198. 95. 53. 134. 144. 232. 81. 104. 59. 246. 297. 258. 229. 275. 281. 179. 200. 200. 173. 180. 84. 121. 161. 99. 109. 115. 268. 274. 158. 107. 83. 103. 272. 85. 280. 336. 281. 118. 317. 235. 60. 174. 259. 178. 128. 96. 126. 288. 88. 292. 71. 197. 186. 25. 84. 96. 195. 53. 217. 172. 131. 214. 59. 70. 220. 268. 152. 47. 74. 295. 101. 151. 127. 237. 225. 81. 151. 107. 64. 138. 185. 265. 101. 137. 143. 141. 79. 292. 178. 91. 116. 86. 122. 72. 129. 142. 90. 158. 39. 196. 222. 277. 99. 196. 202. 155. 77. 191. 70. 73. 49. 65. 263. 248. 296. 214. 185. 78. 93. 252. 150. 77. 208. 77. 108. 160. 53. 220. 154. 259. 90. 246. 124. 67. 72. 257. 262. 275. 177. 71. 47. 187. 125. 78. 51. 258. 215. 303. 243. 91. 150. 310. 153. 346. 63. 89. 50. 39. 103. 308. 116. 145. 74. 45. 115. 264. 87. 202. 127. 182. 241. 66. 94. 283. 64. 102. 200. 265. 94. 230. 181. 156. 233. 60. 219. 80. 68. 332. 248. 84. 200. 55. 85. 89. 31. 129. 83. 275. 65. 198. 236. 253. 124. 44. 172. 114. 142. 109. 180. 144. 163. 147. 97. 220. 190. 109. 191. 122. 230. 242. 248. 249. 192. 131. 237. 78. 135. 244. 199. 270. 164. 72. 96. 306. 91. 214. 95. 216. 263. 178. 113. 200. 139. 139. 88. 148. 88. 243. 71. 77. 109. 272. 60. 54. 221. 90. 311. 281. 182. 321. 58. 262. 206. 233. 242. 123. 167. 63. 197. 71. 168. 140. 217. 121. 235. 245. 40. 52. 104. 132. 88. 69. 219. 72. 201. 110. 51. 277. 63. 118. 69. 273. 258. 43. 198. 242. 232. 175. 93. 168. 275. 293. 281. 72. 140. 189. 181. 209. 136. 261. 113. 131. 174. 257. 55. 84. 42. 146. 212. 233. 91. 111. 152. 120. 67. 310. 94. 183. 66. 173. 72. 49. 64. 48. 178. 104. 132. 220. 57.] DESCR: .. _diabetes_dataset: Diabetes dataset ---------------- Ten baseline variables, age, sex, body mass index, average blood pressure, and six blood serum measurements were obtained for each of n = 442 diabetes patients, as well as the response of interest, a quantitative measure of disease progression one year after baseline. **Data Set Characteristics:** :Number of Instances: 442 :Number of Attributes: First 10 columns are numeric predictive values :Target: Column 11 is a quantitative measure of disease progression one year after baseline :Attribute Information: - Age - Sex - Body mass index - Average blood pressure - S1 - S2 - S3 - S4 - S5 - S6 Note: Each of these 10 feature variables have been mean centered and scaled by the standard deviation times `n_samples` (i.e. the sum of squares of each column totals 1). Source URL: https://www4.stat.ncsu.edu/~boos/var.select/diabetes.html For more information see: Bradley Efron, Trevor Hastie, Iain Johnstone and Robert Tibshirani (2004) "Least Angle Regression," Annals of Statistics (with discussion), 407-499. (https://web.stanford.edu/~hastie/Papers/LARS/LeastAngle_2002.pdf) feature_names: ['age', 'sex', 'bmi', 'bp', 's1', 's2', 's3', 's4', 's5', 's6'] data_filename: C:\Users\tomo\AppData\Local\Programs\Python\Python37-32\lib\site-packages\sklearn\datasets\data\diabetes_data.csv.gz target_filename: C:\Users\tomo\AppData\Local\Programs\Python\Python37-32\lib\site-packages\sklearn\datasets\data\diabetes_target.csv.gz |
データのキーは以下のようになっている。
|
1 2 3 4 5 6 7 |
from sklearn.datasets import load_diabetes ds = load_diabetes() print(ds.keys()) # ddict_keys(['data', 'target', 'DESCR', 'feature_names', 'data_filename', 'target_filename']) |
データの内容
'data'~特徴量データセット
10個の特徴量を列とし、442人の被検者を業とした2次元配列。DESCRに説明されているように、これらのデータは標本平均と標本分散で正規化されており、各特徴量とも、データの和はゼロ(正確には1×10-14~1×10-13のオーダーの実数)、2乗和は1となる。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 |
data: [[ 0.03807591 0.05068012 0.06169621 ... -0.00259226 0.01990842 -0.01764613] [-0.00188202 -0.04464164 -0.05147406 ... -0.03949338 -0.06832974 -0.09220405] [ 0.08529891 0.05068012 0.04445121 ... -0.00259226 0.00286377 -0.02593034] ... [ 0.04170844 0.05068012 -0.01590626 ... -0.01107952 -0.04687948 0.01549073] [-0.04547248 -0.04464164 0.03906215 ... 0.02655962 0.04452837 -0.02593034] [-0.04547248 -0.04464164 -0.0730303 ... -0.03949338 -0.00421986 0.00306441]] |
'target'~糖尿病の進行度
442人に関する10個の特徴量データを測定した1年後の糖尿病の進行度を示す数値。原文でも”a measure of disease progression one year after baseline”としか示されていない。このデータは正規化されていない。
|
1 2 3 4 5 6 7 8 |
target: [151. 75. 141. 206. 135. 97. 138. 63. 110. 310. 101. 69. 179. 185. 118. 171. 166. 144. 97. 168. 68. 49. 68. 245. 184. 202. 137. 85. 131. 283. 129. 59. 341. 87. 65. 102. 265. 276. 252. 90. 100. 55. ..... 72. 140. 189. 181. 209. 136. 261. 113. 131. 174. 257. 55. 84. 42. 146. 212. 233. 91. 111. 152. 120. 67. 310. 94. 183. 66. 173. 72. 49. 64. 48. 178. 104. 132. 220. 57.] |
'feature_names'~特徴名
10種類の特徴量の名称
| sklearn | R | ||
| 0 | age | age | 年齢 |
| 1 | sex | sex | 性別 |
| 2 | bmi | bmi | BMI(Body Mass Index) |
| 3 | bp | map | (動脈の)平均血圧(Average blood pressure) |
| 4 | S1 | tc | 総コレステロール? |
| 5 | S2 | ldl | 悪玉コレステロール(Low Density Lipoprotein) |
| 6 | S3 | hdl | 善玉コレステロール(High Density Lipoprotein) |
| 7 | S4 | tch | 総コレステロール? |
| 8 | S5 | ltg | ラモトリギン? |
| 9 | S6 | glu | 血糖=グルコース? |
scikit-learnでは後半のデータがs1~s6とだけ表示されていて、DESCRにおいても”six blood serum measurements”とだけ書かれている。Rのデータセットでは、これらがtc, ldlなど血清に関する指標の略号で示されている。
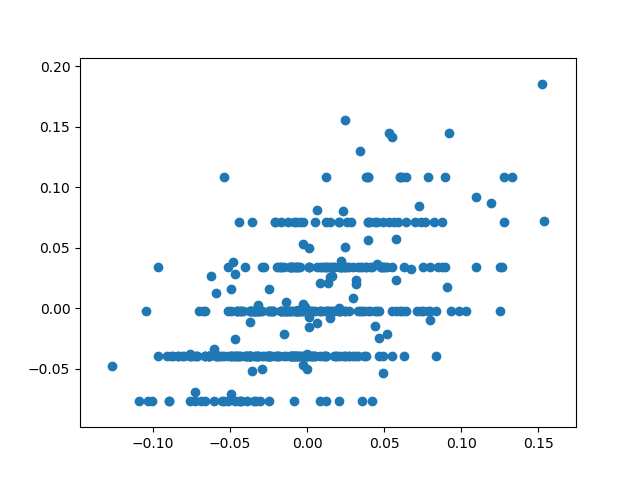
tcとtchはどちらも総コレステロールに関するデータのようだが、どういう違いなのかよくわからない。少なくとも双方に正の相関があるが、ばらつきは大きい。
'filename'~ファイル名
CSVファイルのフルパス名が示されている。scikit-learnの他のデータセットと以下の2点が異なっている。
- 特徴量データdiabetes_data.csvとターゲットデータdiabetes_target.csvの2つのファイルに分かれている
- ファイルの拡張子がcsvとなっているが、区切りはスペースとなっている
|
1 2 3 4 5 |
data_filename: C:...\lib\site-packages\sklearn\datasets\data\diabetes_data.csv.gz target_filename: C:...\lib\site-packages\sklearn\datasets\data\diabetes_target.csv.gz |
diabetes_data.csv
1行に10個の実数がスペース区切りで配置されており、442行のデータがある。442人分の10個の特徴量データ
|
1 2 3 4 5 6 7 |
3.807590643342410180e-02 5.068011873981870252e-02 6.169620651868849837e-02 2.187235499495579841e-02 -4.422349842444640161e-02 -3.482076283769860309e-02 -4.340084565202689815e-02 -2.592261998182820038e-03 1.990842087631829876e-02 -1.764612515980519894e-02 -1.882016527791040067e-03 -4.464163650698899782e-02 -5.147406123880610140e-02 -2.632783471735180084e-02 -8.448724111216979540e-03 -1.916333974822199970e-02 7.441156407875940126e-02 -3.949338287409189657e-02 -6.832974362442149896e-02 -9.220404962683000083e-02 8.529890629667830071e-02 5.068011873981870252e-02 4.445121333659410312e-02 -5.670610554934250001e-03 -4.559945128264750180e-02 -3.419446591411950259e-02 -3.235593223976569732e-02 -2.592261998182820038e-03 2.863770518940129874e-03 -2.593033898947460017e-02 ..... 4.170844488444359899e-02 5.068011873981870252e-02 -1.590626280073640167e-02 1.728186074811709910e-02 -3.734373413344069942e-02 -1.383981589779990050e-02 -2.499265663159149983e-02 -1.107951979964190078e-02 -4.687948284421659950e-02 1.549073015887240078e-02 -4.547247794002570037e-02 -4.464163650698899782e-02 3.906215296718960200e-02 1.215130832538269907e-03 1.631842733640340160e-02 1.528299104862660025e-02 -2.867429443567860031e-02 2.655962349378539894e-02 4.452837402140529671e-02 -2.593033898947460017e-02 -4.547247794002570037e-02 -4.464163650698899782e-02 -7.303030271642410587e-02 -8.141376581713200000e-02 8.374011738825870577e-02 2.780892952020790065e-02 1.738157847891100005e-01 -3.949338287409189657e-02 -4.219859706946029777e-03 3.064409414368320182e-03 |
diabetes_target.csv
ターゲットyに相当する442行の実数データ。
|
1 2 3 4 5 6 7 |
1.510000000000000000e+02 7.500000000000000000e+01 1.410000000000000000e+02 ..... 1.320000000000000000e+02 2.200000000000000000e+02 5.700000000000000000e+01 |
‘DESCR’~データセットの説明
データセットの説明。各特徴量データが標準化されていることが説明されている。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 |
Python - diabetes_01_DESCR.py:5 .. _diabetes_dataset: Diabetes dataset ---------------- Ten baseline variables, age, sex, body mass index, average blood pressure, and six blood serum measurements were obtained for each of n = 442 diabetes patients, as well as the response of interest, a quantitative measure of disease progression one year after baseline. **Data Set Characteristics:** :Number of Instances: 442 :Number of Attributes: First 10 columns are numeric predictive values :Target: Column 11 is a quantitative measure of disease progression one year after baseline :Attribute Information: - Age - Sex - Body mass index - Average blood pressure - S1 - S2 - S3 - S4 - S5 - S6 Note: Each of these 10 feature variables have been mean centered and scaled by the standard deviation times `n_samples` (i.e. the sum of squares of each column totals 1). Source URL: https://www4.stat.ncsu.edu/~boos/var.select/diabetes.html For more information see: Bradley Efron, Trevor Hastie, Iain Johnstone and Robert Tibshirani (2004) "Least Angle Regression," Annals of Statistics (with discussion), 407-499. (https://web.stanford.edu/~hastie/Papers/LARS/LeastAngle_2002.pdf) [Finished in 1.105s] |
データの利用
各データの取得方法
data、targetなどのデータを取り出すのに、以下の2つの方法がある。
- 辞書のキーを使って呼び出す(例:
diabetes['data']) - キーの文字列をプロパティーに指定する(例:
diabetes.data)
dataの扱い
そのまま2次元配列として扱うか、pandas.DataFrameで扱う。特定の特徴量データを取り出すには、ファンシー・インデックスを使う。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 |
from sklearn.datasets import load_diabetes from pandas import DataFrame ds = load_diabetes() df = DataFrame(ds.data, columns=ds.feature_names) print(df[['s1', 's4']]) # s1 s4 # 0 -0.044223 -0.002592 # 1 -0.008449 -0.039493 # 2 -0.045599 -0.002592 # 3 0.012191 0.034309 # 4 0.003935 -0.002592 # .. ... ... # 437 -0.005697 -0.002592 # 438 0.049341 0.034309 # 439 -0.037344 -0.011080 # 440 0.016318 0.026560 # 441 0.083740 -0.039493 |