概要
ここでは、Pythonのscikit-learnパッケージのKNeighborsClassifierクラスにmglearnパッケージのforgeデータを適用してknnの挙動を確認する。
近傍点数を変化させたときのクラス分類の挙動や学習率曲線についてみていく。
近傍点数によるクラス分類の挙動
近傍点数=1の場合
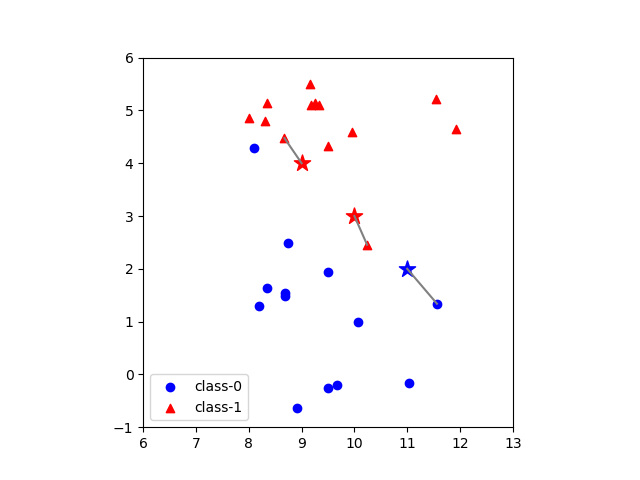
データセットとしてmglearnで提供されているforgeデータを用いて、近傍点数=1とした場合の、3つのテストデータのクラス判定を以下に示す。各テストデータに対して最も距離(この場合はユークリッド距離)が近い点1つが定まり、その点のクラステストデータのクラスとして決定している。
なお、いろいろなところで見かけるforgeデータセットの散布図は当該データセットの特徴量0(横軸)と特徴量1(縦軸)の最小値と最大値に合わせて表示しており、軸目盛の比率が等しくない。ここでは、距離計算に視覚上の齟齬が生じないように、縦軸と横軸の比率を同じとしている。
後の計算のために、このグラフ描画のコードを以下に示す。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 |
import matplotlib.pyplot as plt from sklearn.neighbors import KNeighborsClassifier from mglearn.datasets import make_forge X, y = make_forge() clfr = KNeighborsClassifier(n_neighbors=1) clfr.fit(X, y) col = ['blue', 'red'] test_points = [[9., 4.], [10., 3.], [11., 2.]] nb_dist, nb_idx = clfr.kneighbors(test_points) test_pred = clfr.predict(test_points) fig, ax = plt.subplots() ax.scatter(X[:, 0][y==0], X[:, 1][y==0], marker='o', c=col[0], label="class-0") ax.scatter(X[:, 0][y==1], X[:, 1][y==1], marker='^', c=col[1], label="class-1") ax.legend(loc="lower left") for pts, cls, ids, dists in zip(test_points, test_pred, nb_idx, nb_dist): print(pts) ax.scatter(pts[0], pts[1], marker='*', s=150, c=col[cls]) for id, dst in zip(ids, dists): ax.plot([pts[0], X[id, 0]], [pts[1], X[id, 1]], c='gray') print(" [{:7.4f}, {:7.4f}] - {:7.4f}".format(X[id, 0], X[id, 1], dst)) plt.show() |
概要は以下の通り。
- 5行目で
forgeデータセットを準備 - 7行目で近傍点数を1で指定してクラス分類器を構築
- 8行目で訓練データとして
forgeデータを与える - 12行目で3つのテストデータを準備
- 13行目でテストデータに対する近傍点のインデックスとテストデータまでの距離を獲得
- 14行目でテストデータのクラスを決定
- 18-19行目で訓練データの散布図を描画
- 23行目で、テストデータとそのクラス決定結果、クラス決定に用いられた点群のインデックス、テストデータと各点の距離を並行してループ
- 24行目でテストデータの座標を出力
- 25行目でテストデータを描画
- 26行目のループで、テストデータごとの近傍点に関する処理を実行
- 27行目でテストデータと近傍点の間に直線を描画
- 28行目で近傍点とテストデータからの距離を出力
出力結果は以下の通りで、各予測点に対して近傍点が1つ決定されている。
|
1 2 3 4 5 6 |
[9.0, 4.0] [ 8.6749, 4.4757] - 0.5762 [10.0, 3.0] [10.2403, 2.4554] - 0.5952 [11.0, 2.0] [11.5640, 1.3389] - 0.8689 |
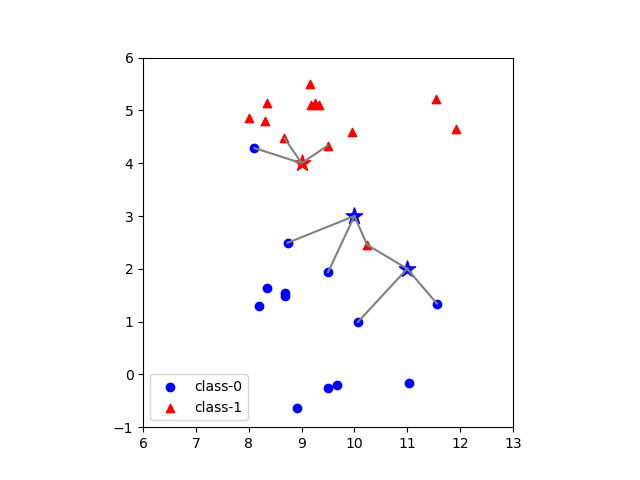
近傍点数=3の場合
先の例で、コードの7行目で近傍点=3で指定してクラス分類器を構築する。
|
1 |
clfr = KNeighborsClassifier(n_neighbors=3) |
一般にknnでは、テストデータに対して複数の近傍点を指定する場合、各近傍点のクラスのうち最も多いものをテストデータのクラスとする(多数決)。
|
1 2 3 4 5 6 7 8 9 10 11 12 |
[9.0, 4.0] [ 8.6749, 4.4757] - 0.5762 [ 9.4912, 4.3322] - 0.5930 [ 8.1062, 4.2870] - 0.9387 [10.0, 3.0] [10.2403, 2.4554] - 0.5952 [ 9.5017, 1.9382] - 1.1729 [ 8.7337, 2.4916] - 1.3645 [11.0, 2.0] [11.5640, 1.3389] - 0.8689 [10.2403, 2.4554] - 0.8858 [10.0639, 0.9908] - 1.3765 |
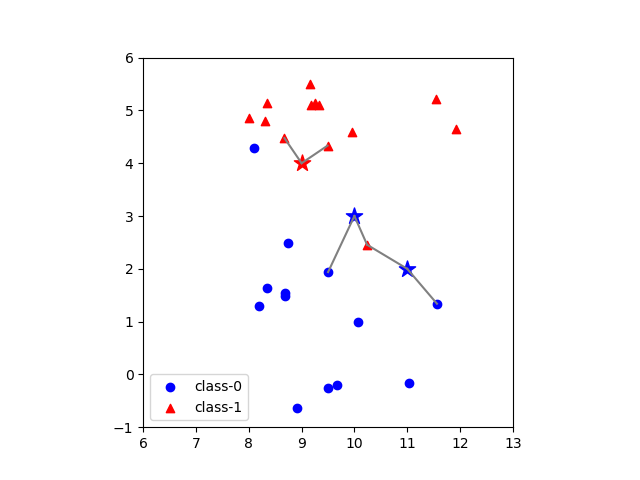
近傍点数=2の場合
テストデータのクラスを近傍点のクラスの多数決で求めるとすると、近傍点数が偶数の時の処理が問題になる。KNeighborsClassifierの場合、偶数でクラス分類が拮抗する場合は、クラス番号が最も小さいものに割り当てられるらしい。実際、n_neighbors=2としたときの3つのテストデータのうち中央の点(10.0, 3.0)については、赤い点(10.24, 2.45)~class-1~距離0.5952の方が青い点(9.5017, 1.9382)~class-0~距離1.1729よりも距離は近いがクラス番号が0である青い点のクラスで判定されている。
|
1 2 3 4 5 6 7 8 9 |
[9.0, 4.0] [ 8.6749, 4.4757] - 0.5762 [ 9.4912, 4.3322] - 0.5930 [10.0, 3.0] [10.2403, 2.4554] - 0.5952 [ 9.5017, 1.9382] - 1.1729 [11.0, 2.0] [11.5640, 1.3389] - 0.8689 [10.2403, 2.4554] - 0.8858 |
偶数の点で多数決で拮抗した場合には、最も近い点のクラスで決定する、平均距離が近い方のクラスで決定するといった方法が考えられるが、この場合は必ず番号が小さなクラスが選ばれるため、若干結果に偏りがでやすいのでは、と考える。
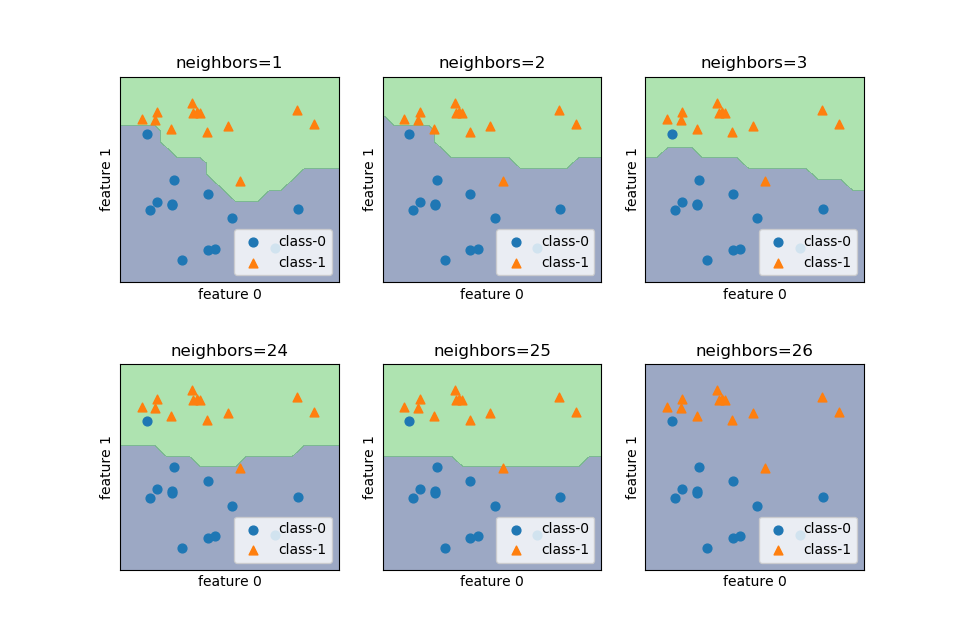
決定境界
近傍点の数を変えた時の決定境界の変化を確認する。k近傍法はscikit-learnのKNeighborsClassifierクラスを利用する。
近傍点の数を1, 2, 3, …と変化させたときの決定境界の変化は以下の通り。
近傍点数が少ないときは訓練データにフィットするよう決定境界が複雑になるが、近傍点数が多いと決定境界は滑らかになる。特に近傍点数が訓練データの点数に等しいとき、全訓練データの多数決でクラス決定され、全領域で判定結果が同じとなる(この場合は近傍点数26が偶数なので、クラス番号の小さいclass-0で決定されている)。
この図を描画したコードを以下に示す。
- 7行目、引数で与えた
Axesに対して決定境界を描く関数を定義- 18行目、決定境界を
contourf()を利用して描いている
- 18行目、決定境界を
- 21行目、引数で与えた
Axesに対してクラスごとに色分けした散布図を描く関数を定義 - 54行目、2次元配列の
Axesを1次元配列として扱っている
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 |
import numpy as np import matplotlib.pyplot as plt from mglearn.datasets import make_forge from sklearn.neighbors import KNeighborsClassifier def draw_decision_boundary(ax, n_neighbors, X, y, X0_field, X1_field): clsfr = KNeighborsClassifier(n_neighbors=n_neighbors) clsfr.fit(X, y) y_predicted = np.empty((len(X1_field), len(X0_field))) for row, x1 in enumerate(X1_field): for col, x0 in enumerate(X0_field): y_predicted[row, col] = clsfr.predict(np.array([[x0, x1]])) ax.contourf(X0_field, X1_field, y_predicted, levels=1, alpha=0.5) def draw_scatter(ax, X0, X1, xlim, ylim): ax.scatter(X0[y==0], X1[y==0], marker='o', s=40, label="class-0") ax.scatter(X0[y==1], X1[y==1], marker='^', s=40, label="class-1") ax.set_xlim(xlim[0], xlim[1]) ax.set_ylim(ylim[0], ylim[1]) ax.set_xlabel("feature 0") ax.set_ylabel("feature 1") ax.tick_params(labelbottom=False, labelleft=False) ax.tick_params(bottom=False, left=False) ax.legend(loc='lower right') X, y = make_forge() X0_scatter = X[:, 0] X1_scatter = X[:, 1] n_X0_field, n_X1_field = 20, 20 y_predicted = np.empty((n_X1_field, n_X0_field)) xlim = (7.5, 12.5) ylim = (-1.5, 6.5) X0_field = np.linspace(xlim[0], xlim[1], n_X0_field) X1_field = np.linspace(ylim[0], ylim[1], n_X1_field) fig, axs = plt.subplots(2, 3, figsize=(9.6, 6.4)) fig.subplots_adjust(hspace= 0.4) n_neighbors_list = [1, 2, 3, 24, 25, 26] axs_1d = axs.reshape(1, -1)[0] for n_neighbors, ax in zip(n_neighbors_list, axs_1d): ax.set_title("neighbors={}".format(n_neighbors)) draw_decision_boundary(ax, n_neighbors, X, y, X0_field, X1_field) draw_scatter(ax, X0_scatter, X1_scatter, xlim, ylim) plt.show() |