概要
irisデータは3つのアヤメの種類(setosa, versicolor, varginica)の150個体について、萼(sepal)と花弁(petal)の長さと幅の組み合わせ4つの特徴量のデータを提供する。これらについて一般的なグラフによる可視化によって俯瞰してみる。
特徴量の分布
クラス分けしない場合
まずアヤメの150個体における4つの特徴量について、3つの種類を区別せずにその分布を見てみる。
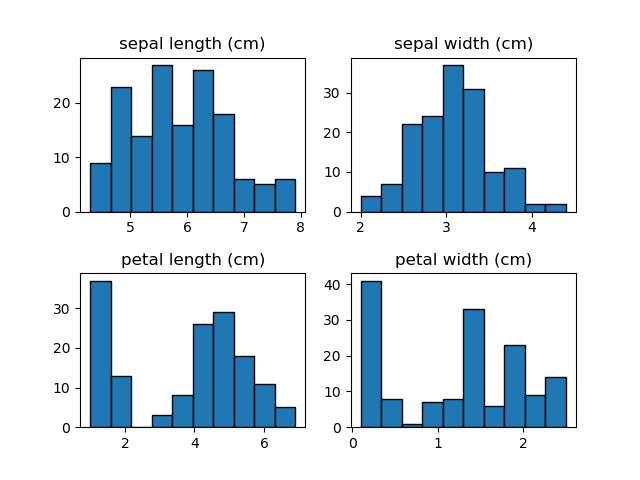
この結果を見る限り特に際立った特徴は見いだせない。敢えて言うなら、萼の長さは若干ばらつきが大きく、萼の幅は割合”きれいな”分布。花弁については、長さ・幅とも値の小さいところで独立した分布が見られる。
このデータが異なる種類のものが混在したものだと知っていれば、花弁の独立した分布は特定の種類のものかもしれないと推測できるくらい。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 |
import numpy as np import matplotlib.pyplot as plt from sklearn.datasets import load_iris iris_data = load_iris() feature_names = iris_data['feature_names'] X = iris_data['data'] n_data, n_features = X.shape fig, axs = plt.subplots(2, 2, figsize=(6.4, 4.8)) ax_1d = [ax for row in axs for ax in row] fig.subplots_adjust(hspace=0.4) for feature in range(n_features): ax_1d[feature].set_title(feature_names[feature]) ax_1d[feature].hist(X[:, feature], ec='k') plt.show() |
クラス分けした場合
次に4つの特徴量について、3つの種類ごとに分けて表示してみる。
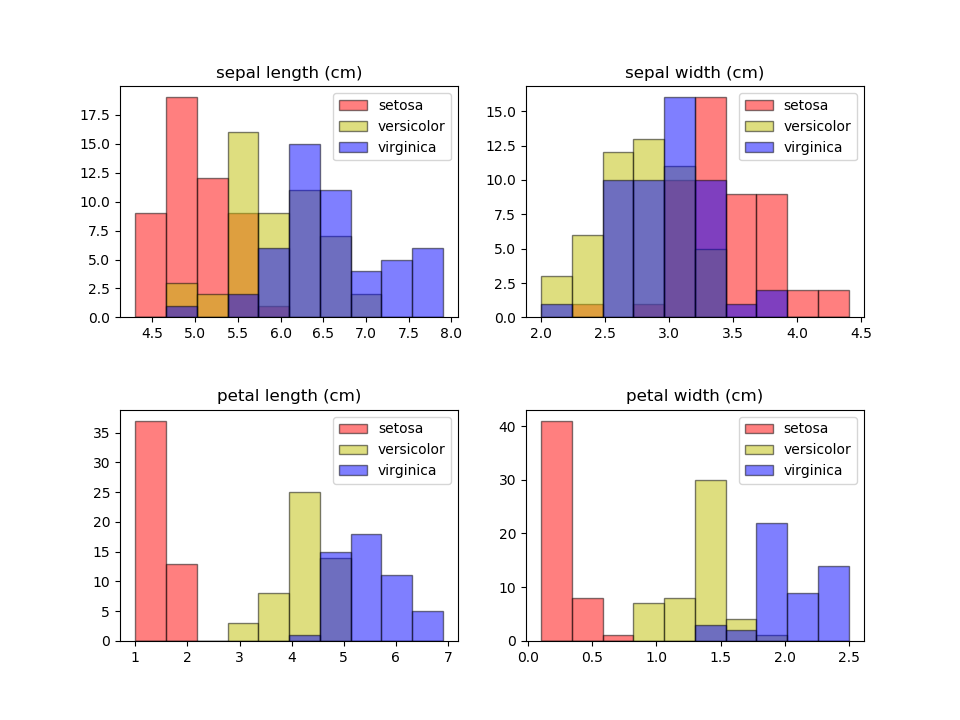
こうすると少し特徴が見えてくる。
花弁の独立した分布はsetosa(ヒオウギアヤメ)のものであることがわかり、額の長さの分布がばらついているのは、複数の種類の特徴量が少しずつずれて重なっているからだということもわかる。
この分布だけだと、花弁の長さ2.5cm、花弁の幅が0.7~0.8cmあたりから小さいと、アヤメの種類はsetosaと特定できそうだが、versicolorとvirginicaは重なっていて、花弁の幅が1.75cmあたりで分けると少し誤判定はあるが概ね分けられそうである。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 |
import numpy as np import matplotlib.pyplot as plt from sklearn.datasets import load_iris iris_data = load_iris() feature_names = iris_data['feature_names'] species_names = iris_data['target_names'] X = iris_data['data'] y = iris_data['target'] n_data, n_features = X.shape species = np.sort(np.array(list(set(y)))) fig, axs = plt.subplots(2, 2, figsize=(9.6, 7.2)) ax_1d = [ax for row in axs for ax in row] fig.subplots_adjust(hspace=0.4) colors = ['r', 'y', 'b'] for feature in range(n_features): ax_1d[feature].set_title(feature_names[feature]) feature_data = X[:, feature] range_max = np.max(feature_data) range_min = np.min(feature_data) for sp in species: ax_1d[feature].hist(feature_data[y==sp], range=(range_min, range_max), bins=10, color=colors[sp], ec='k', alpha=0.5, label=species_names[sp]) ax_1d[feature].legend() plt.show() |
2つの特徴量同士の関係
比較例
例として、萼の長さと萼の幅、萼の長さと花弁の幅、それぞれの間の関係をプロットしてみる。
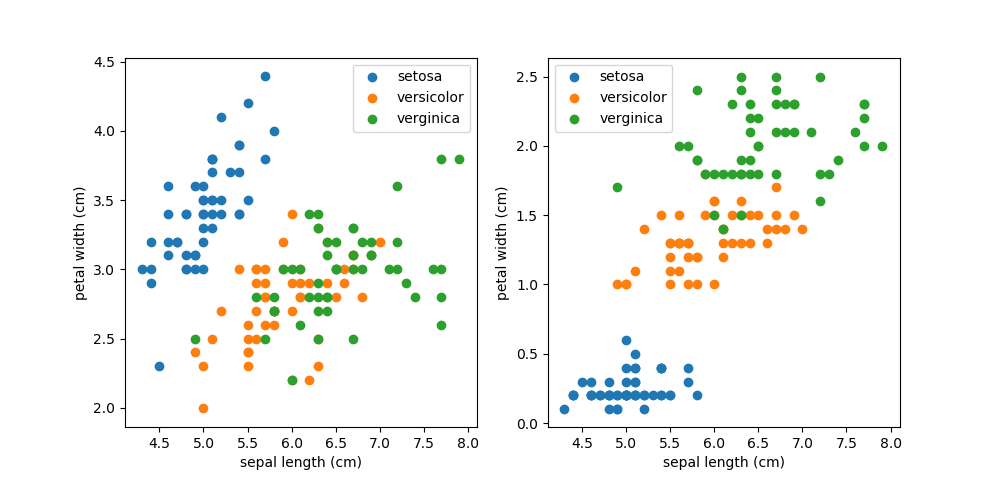
萼の長さと花弁の長さの関係を見ると、setosaは明らかに独立したグループだが、versicolorとverginicaは混ざり合っていて分離できそうにない。先ほどのヒストグラムでは、萼の長さ、萼の幅それぞれだけではversicolorとvirginicaは区分できなかった。2次元でプロットするとそれらがうまく区分する可能性もあるが、この場合はうまくいかないようである。
一方、萼の長さと花弁の長さの関係を比べると、versicolorとversinicaも何とか区分できそうである。よくみると、この3つの区分は萼の長さと関係なく、花弁の幅のみで概ね区分できそうである。これも先ほどの花弁の幅のヒストグラムの結果と符合する。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 |
import numpy as np import matplotlib.pyplot as plt from sklearn.datasets import load_iris iris_data = load_iris() X = iris_data['data'] y = iris_data['target'] data_setosa = X[y==0] data_versicolor = X[y==1] data_verginica = X[y==2] sl, sw, pl, pw = (0, 1, 2, 3) fig, axs = plt.subplots(1, 2, figsize=(10, 4.8)) a, b = (sl, sw) axs[0].scatter(data_setosa[:, a], data_setosa[:, b], label="setosa") axs[0].scatter(data_versicolor[:, a], data_versicolor[:, b], label="versicolor") axs[0].scatter(data_verginica[:, a], data_verginica[:, b], label="verginica") a, b = (sl, pw) axs[1].scatter(data_setosa[:, a], data_setosa[:, b], label="setosa") axs[1].scatter(data_versicolor[:, a], data_versicolor[:, b], label="versicolor") axs[1].scatter(data_verginica[:, a], data_verginica[:, b], label="verginica") for ax in axs: ax.set_xlabel(iris_data.feature_names[a]) ax.set_ylabel(iris_data.feature_names[b]) ax.legend() plt.show() |
scatter_matrixによる確認
上記のような特徴量の組み合わせは、特徴量がn個の場合にはnC2通りとなる。irisデータの場合、特徴量は4つだから6個の特徴量ペアがあり得る。pandasのscatter_matrixを利用すると、このような特徴量のペアについて網羅的に確認できる。
ただしscatter_matrixでは、対角要素のヒストグラムを特徴量ごとに分けることはできないようだ。
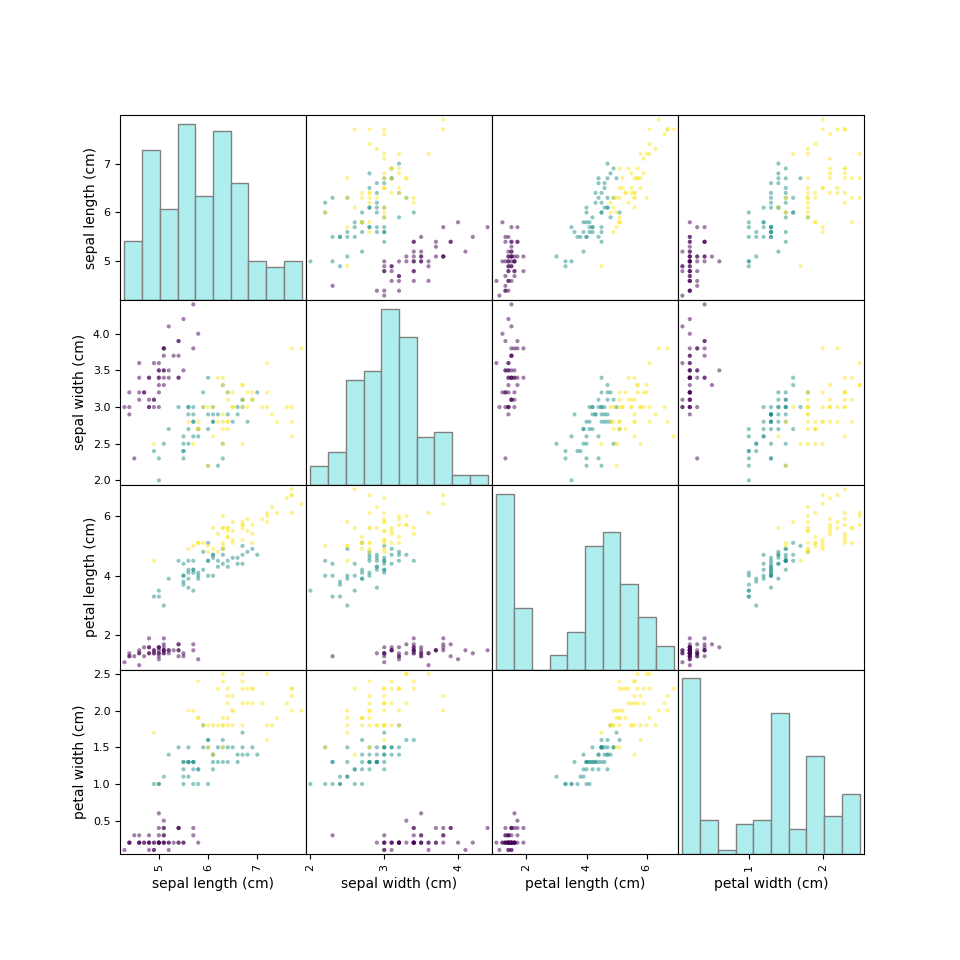
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 |
import pandas as pd import matplotlib.pyplot as plt from sklearn.datasets import load_iris iris_dataset = load_iris() iris_dataframe = pd.DataFrame(iris_dataset.data, columns=iris_dataset.feature_names) pd.plotting.scatter_matrix(iris_dataframe, figsize=(9.6, 9.6), c=iris_dataset.target, hist_kwds={'ec':'gray', 'color':'paleturquoise'}) plt.show() |
pairplotによる確認
seabornのpairplotを使うと、対角要素に各特徴量ごとの頻度分布/密度分布を表示することができる。pairplotの場合、ターゲットの品種を文字列で与えるとそれに従った色分けをしてくれて、対角要素の密度分布も品種ごとに分けてくれる。
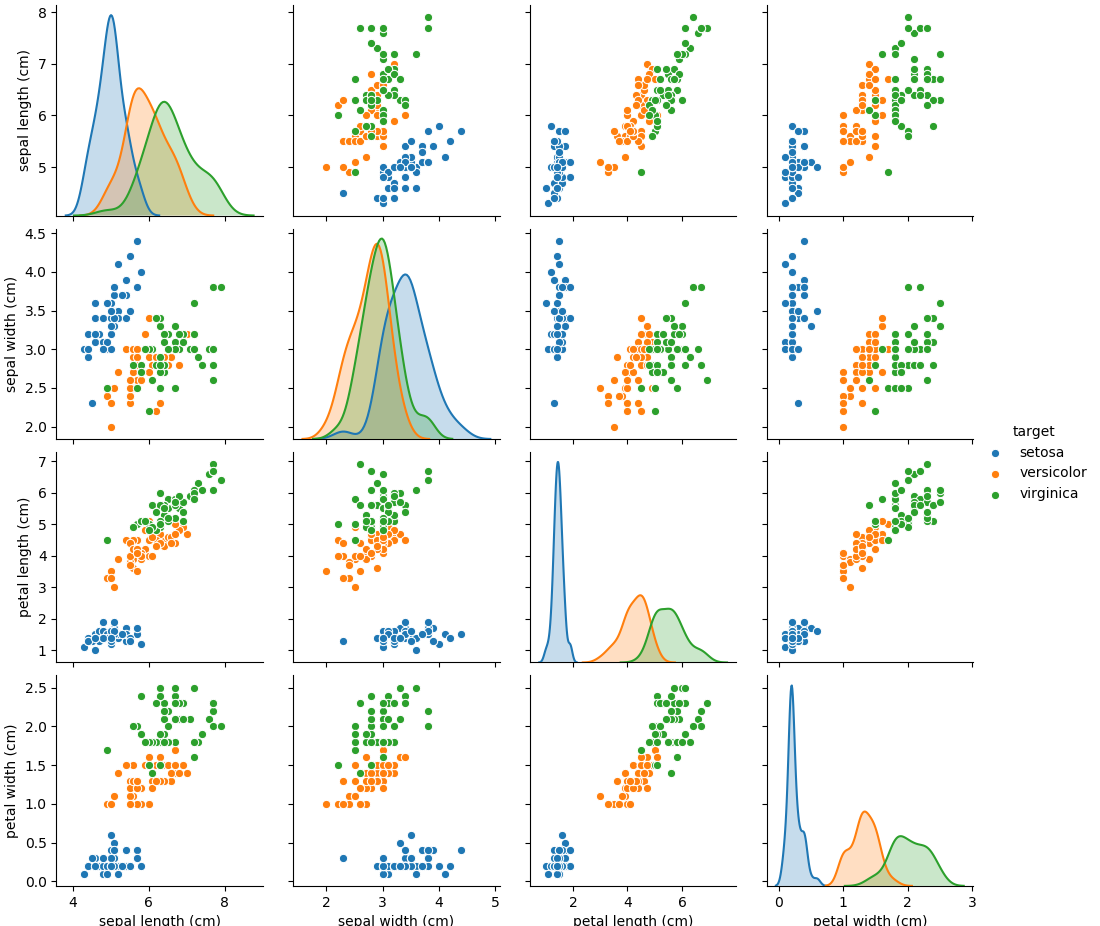
ペアプロットの結果から、3つの種類は複数の散布図で比較的きれいにグループとなっていることがわかる。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 |
import pandas as pd import matplotlib.pyplot as plt import seaborn as sns from sklearn.datasets import load_iris iris_ds = load_iris() df = pd.DataFrame(iris_ds.data, columns=iris_ds.feature_names) df['target'] = iris_ds.target df.loc[df['target']==0, 'target'] = "setosa" df.loc[df['target']==1, 'target'] = "versicolor" df.loc[df['target']==2, 'target'] = "virginica" g = sns.pairplot(df, hue='target') g.fig.set_figheight(9.6) g.fig.set_figwidth(11) plt.show() |
3つの特徴量の関係
最後に、4つの特徴量のうち3つを取り出して3次元の散布図で表示してみる。2次元の散布図ではversicolorとvirginicaで若干の重なりがあるが、3次元化するときれいに分かれるかもしれない。
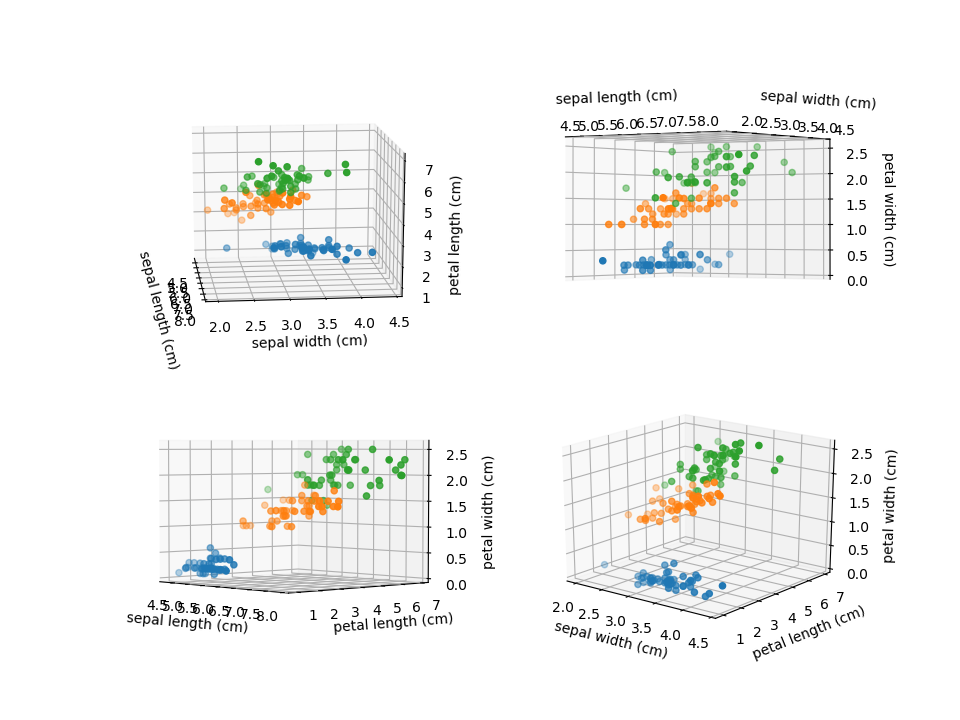
3次元空間で見ても若干の重なりはあるが、2つの特徴量だけの時に比べて、よりグループ分離の精度が高まることは期待できそうだ。
考えてみれば、アヤメの品種区分のように特徴量が少ない場合のクラス分類問題は、1次元の頻度分布、2次元・3次元の頻度分布のように次元を増やして確認ができれば、区分は比較的容易なように思われる。一方で人の間隔では3次元を認識するのがやっとなので、特徴量の数が増えた時には太刀打ちできない。
畢竟、機械学習・AIとは人間が認識制御困難な多数の特徴量=多次元における判別や相関を如何に実行するかというところなのでは、と思われる。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 |
import numpy as np import matplotlib.pyplot as plt from mpl_toolkits.mplot3d import Axes3D from sklearn.datasets import load_iris iris_ds = load_iris() X = iris_ds.data y = iris_ds.target feature_names = iris_ds.feature_names sl, sw, pl, pw = (0, 1, 2, 3) species = (0, 1, 2) combinations = np.array([ [sl, sw, pl], [sl, sw, pw], [sl, pl, pw], [sw, pl, pw] ]) fig = plt.figure(figsize=(9.6, 7.2)) ax1 = fig.add_subplot(221, projection='3d') ax2 = fig.add_subplot(222, projection='3d') ax3 = fig.add_subplot(223, projection='3d') ax4 = fig.add_subplot(224, projection='3d') axs = [ax1, ax2, ax3, ax4] for ax, comb in zip(axs, combinations): f0, f1, f2 = comb[0], comb[1], comb[2] xs, ys, zs = X[:, f0], X[:, f1], X[:, f2] for sp in species: ax.scatter(xs[y==sp], ys[y==sp], zs[y==sp]) ax.set_xlabel(feature_names[f0]) ax.set_ylabel(feature_names[f1]) ax.set_zlabel(feature_names[f2]) plt.show() |