概要
k-最近傍法(k nearest neighbors: knn)によるクラス分類は、テストデータの近傍の訓練データからテストデータのクラスを決定する。その手法は単純で、特段の学習処理はせず、訓練データの特徴量とクラスを記憶するのみで、テストデータが与えられたときに近傍点からクラスを決定する。手順は以下の通り。
- 特徴量とクラス分類の訓練データセットを記憶する
- テストデータが与えられたら、特徴量空間の中で近傍点を選ぶ
- 近傍点のクラスからテストデータのクラスを決定する
パラメーターは近傍点の数で、1以上の自然数を設定できる。
利用方法
scikkit-learnのKNeighborsClassifierクラスの利用方法は以下の通り。
sklearn.neighborsからKNeighborsClassifierをインポート- コンストラクターの引数に近傍点数
n_neighborsを指定して、KNeighborsClassifierのインスタンスを生成 fit()メソッドに訓練データの特徴量と属性値を与えて学習predict()メソッドにテストデータの特徴量を指定して、属性値を予測- 必要に応じて、
kneighbors()メソッドでテストデータの近傍点情報を取得
コンストラクターには、通常n_neighborsで近傍点を指定する。デフォルトはn_neighbors=5。
- KNeighborsClassifier(n_neighbors=n)
- nは近傍点の数。この他の引数に、近傍点を発見するアルゴリズムなどが指定できるようだ。
fit()メソッドに与える訓練データは、特徴量セットと属性値の2つ。
fit(X, y)Xは訓練データセットの特徴量データで、データ数×特徴量数の2次元配列。yは訓練データセットのクラスデータで要素数はデータ数に等しい
テストデータの属性値の予測は、predict()メソッドにテストデータの特徴量を与える。
y = predict(X)Xはテストデータの特徴量データで、データ数×特徴量数の2次元配列。戻り値yは予測されたクラスデータで要素数はデータ数に等しい。
テストデータに対する近傍点の情報を、kneighbors()メソッドで得ることができる。
neigh_dist, neigh_ind = kneighbors(X)- X_testはテストデータの特徴量データで、データ数×特徴量数の2次元配列。戻り値y_testは予測された属性値データで要素数はデータ数に等しい。
neigh_dist, neigh_ind = kneighbors(X)- テストデータの特徴量
Xを引数に与え、近傍点に関する情報を得る。neigh_distは各テストデータから各近傍点までの距離、neigh_indは各テストデータに対する各近傍点のインデックス。いずれも2次元の配列で、テストデータ数×近傍点数の2次元配列となっている。
実行例
以下の例では、n_neighbors=2としてKNeighborsClassifierのインスタンスを準備している。
これに対してfit()メソッドで、2つの特徴量とそれに対するクラス値を持つ訓練データを6個与えている。特徴量データX_trainは行数がデータ数、列数が特徴量の数となる2次元配列を想定している。また属性値y_trainは訓練データ数と同じ要素数の1次元配列。
| 特徴量1 | 特徴量2 | クラス値 |
| -2 | -1 | 0 |
| -1 | -2 | 0 |
| -0.5 | -0.5 | 0 |
| 0.5 | 0.5 | 1 |
| 1 | 2 | 1 |
| 2 | 1 | 1 |
これらの訓練データに対して、テストデータの特徴量X_testとして(-0.5, -1.5)、(1, 0)の2つを与えた時の出力を見てみる。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 |
import numpy as np from sklearn.neighbors import KNeighborsClassifier X_train = np.array([ [-2, -1], [-1, -2], [-0.5, -0.5], [0.5, 0.5], [1, 2], [2, 1] ]) y_train = np.array([0, 0, 0, 1, 1, 1]) clf = KNeighborsClassifier(n_neighbors=3) clf.fit(X_train, y_train) X_test = np.array([[-0.5, -1.5], [1, 0]]) y_pred = clf.predict(X_test) neigh_dist, neigh_ind = clf.kneighbors(X=X_test) print("X_train=\n{}".format(X_train)) print("y_train={}".format(y_train)) print("X_test=\n{}".format(X_test)) print("y_pred={}".format(y_pred)) print("neighbors' distance=\n{}".format(neigh_dist)) print("neighbors' indicies=\n{}".format(neigh_ind)) |
このコードの実行結果は以下の通り。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 |
X_train= [[-2. -1. ] [-1. -2. ] [-0.5 -0.5] [ 0.5 0.5] [ 1. 2. ] [ 2. 1. ]] y_train=[0 0 0 1 1 1] X_test= [[-0.5 -1.5] [ 1. 0. ]] y_pred=[0 1] neighbors' distance= [[0.70710678 1. 1.58113883] [0.70710678 1.41421356 1.58113883]] neighbors' indicies= [[1 2 0] [3 5 2]] |
属性値の予測結果については、2つのテストデータに対して2つのクラス値0と1が返されている。
kneighbors()メソッドの戻り値から、1つ目のテストデータにはインデックスが1, 2, 0の3つの点とそれぞれへの距離0.7071, 1, 1.5811が、2つ目のテストデータにはインデックスが3, 5, 2の点とそれぞれへの距離0.7071, 1.4142, 1.5811が得られる。
- 1つ目のテストデータ
(-0.5, -1.5)からの距離X_train[1]=(-1, -2)→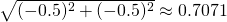
X_train[2]=(-0.5, -0.5)→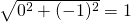
X_train[0]=(-2, -1)→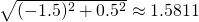
- 2つ目のテストデータ
(1, 0)からの距離X_train[3]=(0.5, 0.5)→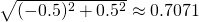
X_train[5]=(2, 1)→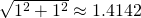
X_train[2]=(-0.5, -0.5)→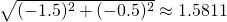
y_predは、テストデータごとに2つの近傍点のクラス値から多数決でクラス値を決定している。
- 1つ目のテストデータの属性値
y_train[1]=0、y_train[2]=0、y_train[0]=0の多数決→0
- 2つ目のテストデータの属性値
y_train[3]=1、y_train[5]=1、y_train[2]=0の多数決→1
この様子を特徴量平面上に描いたのが以下の図である。各点の色は、各データのクラスを示していて、下方の点は3つの近傍点のクラスが全て0なのでテストデータのクラスも0、右方の点は近傍点のうち2つがクラス1で1つがクラス0なのでテストデータのクラスは多数決で1となっている様子がわかる。
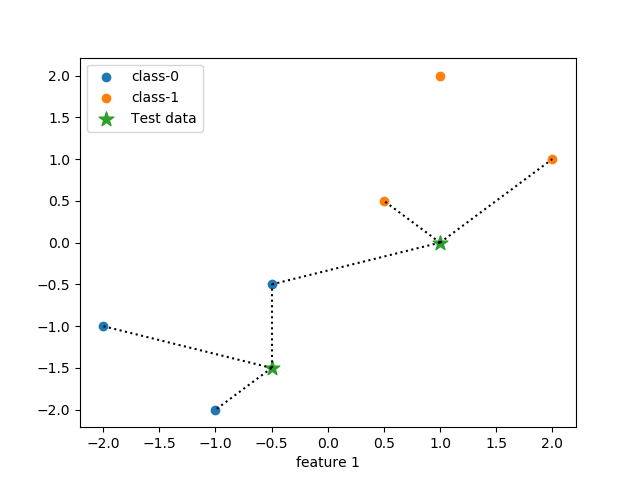
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 |
import numpy as np import matplotlib.pyplot as plt from sklearn.neighbors import KNeighborsClassifier X_train = np.array([ [-2, -1], [-1, -2], [-0.5, -0.5], [0.5, 0.5], [1, 2], [2, 1] ]) y_train = np.array([0, 0, 0, 1, 1, 1]) clf = KNeighborsClassifier(n_neighbors=3) clf.fit(X_train, y_train) X_test = np.array([[-0.5, -1.5], [1, 0]]) y_pred = clf.predict(X_test) neigh_dist, neigh_ind = clf.kneighbors(X=X_test) fig, ax = plt.subplots() X0 = X_train[y_train==0] X1 = X_train[y_train==1] ax.scatter(X0[:, 0], X0[:, 1], label="class-0") ax.scatter(X1[:, 0], X1[:, 1], label="class-1") ax.scatter(X_test[:, 0], X_test[:, 1], marker='*', s=120, label="Test data") for tests, ind in zip(X_test, neigh_ind): for neigh in ind: ax.plot( [tests[0], X_train[neigh][0]], [tests[1], X_train[neigh][1]], color='k', linestyle='dotted') ax.set_xlabel("feature 0") ax.set_xlabel("feature 1") ax.legend(loc='upper left') plt.show() |
各種データに対する適用例